seekparentf90을 이용한 친자감정과 부모 찾기는 이미 포스팅한 적이 있다.
2020.02.12 - [Animal Breeding/Genomic Selection] - SNP 유전체 자료를 이용한 아비 어미 찾기
SNP 유전체 자료를 이용한 아비 어미 찾기
imputation 또는 qc 과정에서, 사용하는 프로그램에 따라 친자감정 결과를 제시하거나 또는 진짜 부모라고 여겨지는 개체를 찾아 주기도 한다. 여기서는 아비, 어미를 본격적으로 찾아주는 프로그
bhpark.tistory.com
qcf90 또는 pregsf90 프로그램을 통해서 대부분의 SNP genotype quality control을 수행할 수 있다. 그러나 이런 프로그램들은 여러 버전의 마이크로 어레이를 동시에 이용하여 quality control을 할 수 없고, 하려 한다면 imputation을 해야 한다. imputation이 필요하다면 imputation을 해서 quality control을 하면 되나, raw data를 그대로 이용하여 친자감정을 하거나 부모를 찾으려 하는 경우도 있다. seekparentf90은 약간(?)의 자료 가공을 거쳐 imputation 하지 않고, 친자 감정하거나 부모를 찾을 수 있는 기능을 제공하고 있다.
먼저 BovineSNP50V2, BovineSNP50V3, Hanwoo50V1의 GenomeStudio에서 출력한 finalreport를 illumina2pregs 프로그램을 이용하여 coded하였다고 하자. 그럴 경우 각 버전의 genotype과 map file은 다음과 같다.
BovineSNP50V2
- genotype
- map file

BovineSNP50V3
- genotype
- map file
Hanwoo50V1
- genotype
- map file
먼저 map file을 합쳐야 한다.
세 개의 map file을 읽어서 seekparentf90을 위한 map file 만드는 SAS 프로그램
/*
file name : make_snpmapfile_for_seekparentf90.sas
seekparentf90 프로그램을 위한 map file 만들기
BovineSNP50V2, BovineSNP50V3, HanwooSNP50V1 map file을 하나로 합치기
*/
/* BovineSNP50V2 snp_map 파일 읽기 */
data a1 ;
infile 'snp_map_bv2' firstobs = 2;
informat name $37. ;
informat bv2chrmo best32. ;
informat bv2pos best32. ;
informat bv2oindex best32. ;
format name $37. ;
format bv2chrmo best32. ;
format bv2pos best32. ;
format bv2oindex best32. ;
input
name
bv2chrmo
bv2pos
bv2oindex
;
bv2index = _n_;
run;
/* BovineSNP50V3 snp_map 파일 읽기 */
data b1 ;
infile 'snp_map_bv3' firstobs = 2;
informat name $37. ;
informat bv3chrmo best32. ;
informat bv3pos best32. ;
informat bv3oindex best32. ;
format name $37. ;
format bv3chrmo best32. ;
format bv3pos best32. ;
format bv3oindex best32. ;
input
name
bv3chrmo
bv3pos
bv3oindex
;
bv3index = _n_;
run;
/* HanwooSNP50V1 snp_map 파일 읽기 */
data c1 ;
infile 'snp_map_hv1' firstobs = 2;
informat name $37. ;
informat hv1chrmo best32. ;
informat hv1pos best32. ;
informat hv1oindex best32. ;
format name $37. ;
format hv1chrmo best32. ;
format hv1pos best32. ;
format hv1oindex best32. ;
input
name
hv1chrmo
hv1pos
hv1oindex
;
hv1index = _n_;
run;
/* bv2, bv3, hv1 머지하기 */
/* sort */
proc sort data = a1;
by name;
run;
proc sort data = b1;
by name;
run;
proc sort data = c1;
by name;
run;
/* 머지하기 */
data t1;
merge a1 b1 c1;
by name;
run;
/* 이름은 같은데 염색체, 위치가 다른 SNP */
/* 있으면 안된다. 있다면 아마도 genome build가 칩마다 다른 것일 수도 */
data t1_e1;
set t1;
/* bv2와 bv3 비교 */
if bv2chrmo ~= . and bv3chrmo ~= . and bv2chrmo ~= bv3chrmo then output;
/* bv3와 hv1 비교 */
if bv3chrmo ~= . and hv1chrmo ~= . and bv3chrmo ~= hv1chrmo then output;
run;
/* index, 염색체 번호, 위치 번호 정리 */
data t2;
set t1;
/* 없었던 SNP index는 0 */
if bv2index = . then bv2index = 0;
if bv3index = . then bv3index = 0;
if hv1index = . then hv1index = 0;
/* 각 SNP의 염색체 번호 */
chr = bv2chrmo;
if bv2chrmo = . then chr = bv3chrmo;
if bv2chrmo = . and bv3chrmo = . then chr = hv1chrmo;
/* 각 SNP의 위치 */
pos = bv2pos;
if bv2pos = . then pos = bv3pos;
if bv2pos = . and bv3pos = . then pos = hv1pos;
run;
/* dataset을 열어서 확인 */
proc sort data = t2;
by chr pos;
run;
data _null_;
set t2;
file 'mapfile_for_seekparentf90.txt';
if _n_ = 1 then put "SNP_ID Chr Pos chip1 chip2 chip3";
put name @41 chr @51 pos @61 bv2index @71 bv3index @81 hv1index;
run;
결과적으로 만들어진 map file은 다음과 같다.
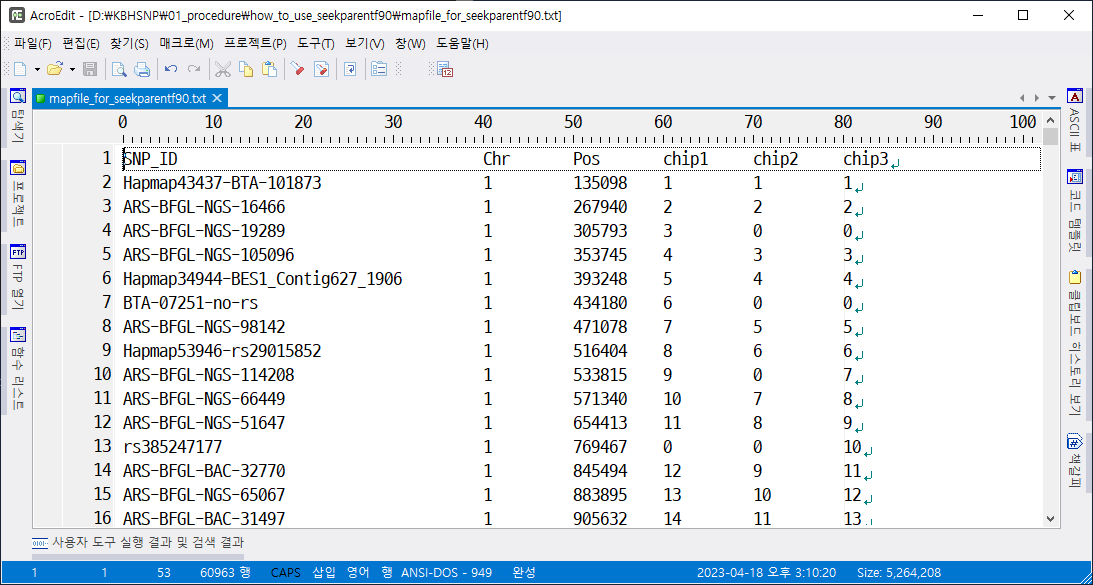
각 genotype 파일에 chip number를 넣어야 한다.
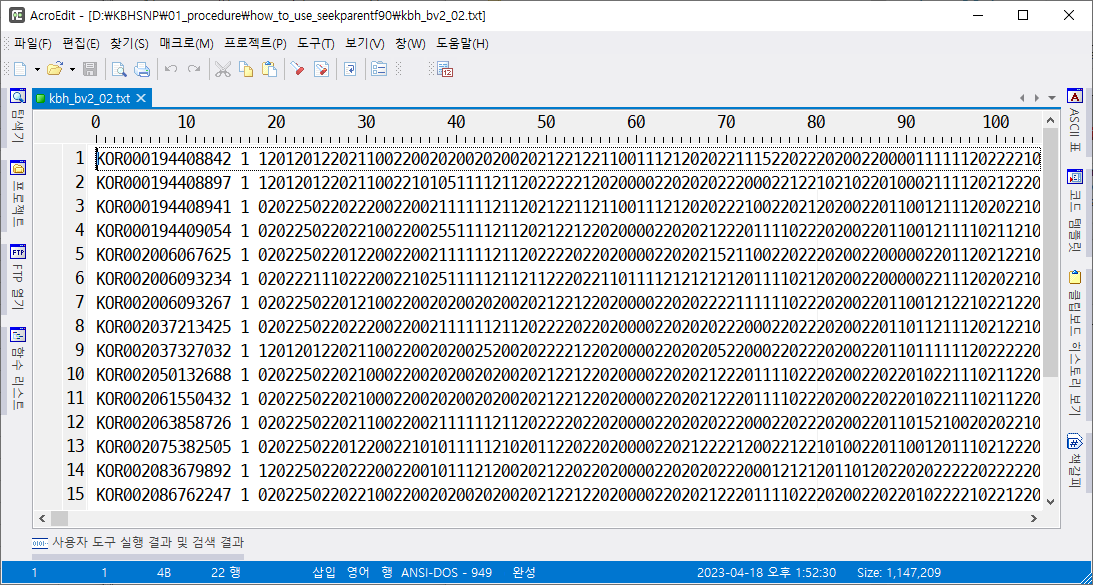
BovineSNP50V2에 chip number 1을 넣는 awk 프로그램은 다음과 같다. insert_chip_number_bv2.awk로 저장한다.
BEGIN{
}
{
print $1 " 1 " $2 > "kbh_bv2_02.txt"
}
END {
}
위 awk 프로그램을 실행하는 방법은 다음과 같다.
awk -f insert_chip_number_bv2.awk kbh_bv2.txt
pause
위 내용을 run_insert_chip_number_bv2.awk.bat에 저장하면 더블 클릭하여 실행할 수 있다.
결과적으로 생긴 파일은 다음과 같다.
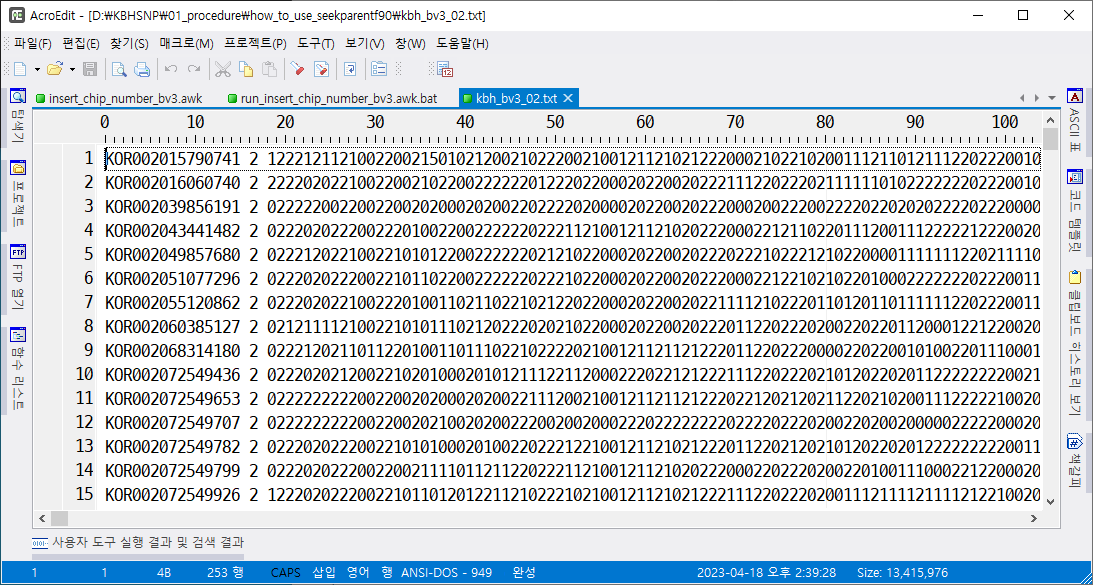
BovineSNPV3 genotype에 chip number 2를 넣는 awk 프로그램
BEGIN{
}
{
print $1 " 2 " $2 > "kbh_bv3_02.txt"
}
END {
}
위 소스를 insert_chip_number_bv3.awk으로 저장
위 awk 프로그램을 실행하는 방법
awk -f insert_chip_number_bv3.awk kbh_bv3.txt
pause
위 명령어를 run_insert_chip_number_bv3.awk.bat에 저장하면 더블 클릭하여 실행할 수 있다.
결과적으로 생긴 파일은 다음과 같다.
HanwooSNPV1 genotype에 chip number 3을 넣는 awk 프로그램
BEGIN{
}
{
print $1 " 3 " $2 > "kbh_hv1_02.txt"
}
END {
}
위 소스를 insert_chip_number_hv1.awk으로 저장
위 awk 프로그램을 실행하는 방법
awk -f insert_chip_number_hv1.awk kbh_hv1.txt
pause
위 명령어를 run_insert_chip_number_hv1.awk.bat에 저장하면 더블 클릭하여 실행할 수 있다.
결과적으로 생긴 파일은 다음과 같다.
위 세 genotype 파일을 하나로 합치는 명령어
cat kbh_bv2_02.txt kbh_bv3_02.txt kbh_hv1_02.txt > kbh_genotype.txt
pause
위 명령어를 make_one_genotype_file.bat으로 저장하면 더블 클릭하여 실행할 수 있다.
결과적으로 생긴 파일은 다음과 같다.
친자감정을 하는 것이므로 개체의 아비와 어미가 누구인지를 나타내는 혈통파일이 있어야 한다. 준비한 혈통 파일은 다음과 같다(kbh_pedigree_yob.txt)
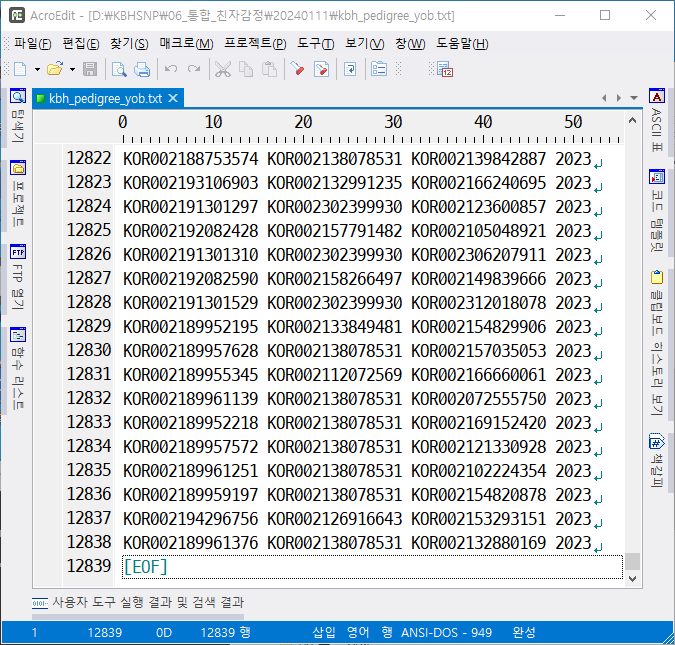
이제 seekparentf90으로 친자감정을 하고, 친자감정 불일치일 경우 아비 또는 어미를 찾아 보자.
seekparentf90을 실행하는 명령어는 다음과 같다.
seekparentf90 --pedfile kbh_pedigree_yob.txt --snpfile kbh_genotype.txt --chips mapfile_for_seekparentf90.txt --yob --seeksire_in_ped --seekdam_in_ped --exclude_chr 30 31 32 33 --seektype 1 --duplicate --full_log_checks | tee seekparentf90_01.log
pause
위 명령어를 run_seekparentf90.bat으로 저장하면 더블클릭으로 실행할 수 있다.
명령어 설명
seekparentf90 명령어
--pedfile kbh_pedigree_yob.txt 혈통 파일. 혈통 파일은 header 없음.
--snpfile kbh_genotype.txt 위에서 준비한 genotype 파일
--chips mapfile_for_seekparentf90.txt 위에서 준비한 map file
--yob 혈통의 넷째 컬럼에 출생연도가 있음
--seeksire_in_ped 혈통에서 아비 찾기
--seekdam_in_ped 혈통에서 어미 찾기
--exclude_chr 30 31 32 33 30에서 33번 염색체는 제외
--seektype 1 불일치인 경우만 아비 및 어미 찾기
--duplicate 유전자형이 중복인 개체들이 있는지 검사
--full_log_checks 많은 로그 기록
| tee seekparentf90_01.log 화면에 프린트되는 것을 파일에도 기록
로그는 다음과 같다.
----------------------------------------------------
| SeekParentf90 |
| Parent-Offspring tests |
| and detection of paternity |
| based on SNP markers |
| |
| 2013 - Version 1.47 |
| (Last update: Dec 15, 2022) |
| |
| |
| INIA Las Brujas, Uruguay |
| University of Georgia, Athens, US |
----------------------------------------------------
Options
--pedfile kbh_pedigree_yob.txt
--snpfile kbh_genotype.txt
--yob
--seeksire_in_ped
--seekdam_in_ped
--seektype 1
--chips mapfile_for_seekparentf90.txt
--exclude_chr 30 31 32 33
--full_log_checks
Using Jenkins hash function
Maximum length for alphanumeric fields: 20
Fields to read: animal, sire, dam, yob (4)
Elapsed time for reading and hash: .250
Pedigree file "kbh_pedigree_yob.txt": 12412 records
Total number of animals in pedigre: 12412
Statistics for Year of Birth
Minimun: 1990
Maximun: 2023
Read multiple Chip SNP file: "kbh_genotype.txt"
Number of SNP from snp_info file: 60961
***** WARNING ***** Number of duplicate SNP by chr_pos: 93
***** List of duplicate SNP: "duplicate_snp_chr_pos"
Exclude Chr(s)
Chr: 30 1190
Chr: 31 11
Chr: 32 13
Chr: 33 775
Chip identification
Chip 1: CHIP1
Chip 2: CHIP2
Chip 3: CHIP3
Number of SNP by Chip
Chip 1: 54609 50908 47514
Chip 2: 0 53218 49760
Chip 3: 0 0 53866
Exclude SNP: 1989
Maximum Available SNP for calculations: 58972
Number of effective SNP by Chip for parent-progeny conflicts
Chip 1: 52886 49233 46109
Chip 2: 0 51278 48090
Chip 3: 0 0 52195
Column position in file for the first marker: 19
Format to read SNP file: (18x,800000i1)
Number of SNPs: 60961
Threshold for exclusions - Maximun number of SNP: 58972
Number of SNP > 130, set the threshold probability to exclude= 1.000
Number of SNP > 130, set the threshold probability to assign = .500
Threshold based on percent of conflicts to exclude: 1.000
Threshold based on percent of conflicts to assign: .500
Number of records in SNP file: 1884
Number of records present in pedigree: 1883 1883
Number of records by Chip
Chip 1: 21
Chip 2: 252
Chip 3: 1611
Call Rate
Min CR: 5346 .90
Max CR: 42 1.00
AVG CR: 668 .99
Number of samples with low call rate ( 0.90 ): 0
Samples with low call rate for effective SNP 0
Check all pedigree for parent-progeny conflicts
Pedigree records with genotype: 1883
Records not tested (low call rate < 0.90 ): 0
Records tested: 1883
Pair parent/progeny tested: 1403
Pair with conflicts: 19 1.4 %
NOT tested (parent low call rate): 0
Sire-progeny tested: 715
Sire-progeny with conflicts: 11 1.5 %
NOT tested (parent low call rate): 0
Dam-progeny tested: 688
Dam-progeny with conflicts: 8 1.2 %
NOT tested (parent low call rate): 0
Seek parent for SIRES
Get list of parents: detected from pedigree
Number of Sires: 171
Seek method: Only for animals with conflicts
Total number of animals: 11
Found: 7
Not Found: 4
Found but cannot assign: 0
Seek parent for DAMS
Get list of parents: detected from pedigree
Number of Dams : 666
Seek method: Only for animals with conflicts
Total number of animals: 8
Found: 3
Not Found: 5
Found but cannot assign: 0
Output files
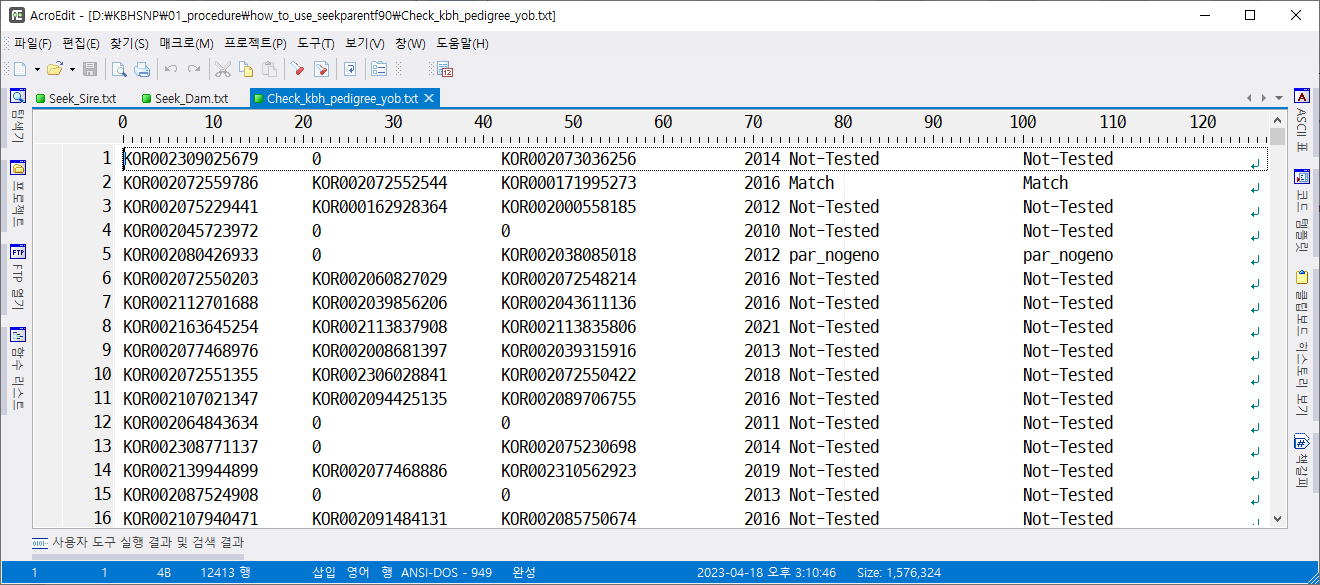
Pedigree file with removed parents for animals with conflicts: "Check_kbh_pedigree_yob.txt"
Full reports
"Parent_Progeny_Conflicts.txt"
"Parent_Progeny_Conflicts_Summary.txt"
"Seek_Sire.txt"
"Seek_Dam.txt"
중간에
***** WARNING ***** Number of duplicate SNP by chr_pos: 93
***** List of duplicate SNP: "duplicate_snp_chr_pos“
로그가 있는데 duplicate_snp_chr_pos 파일을 열어 보면 다음과 같다,
Duplicate SNP chr_pos: 1_59409838 2
Duplicate SNP chr_pos: 13_25606469 2
Duplicate SNP chr_pos: 3_58040470 2
Duplicate SNP chr_pos: 14_27271835 2
Duplicate SNP chr_pos: 29_28647816 2
Duplicate SNP chr_pos: 15_15014275 2
Duplicate SNP chr_pos: 2_50689222 2
Duplicate SNP chr_pos: 11_33265117 2
Duplicate SNP chr_pos: 7_1071389 2
Duplicate SNP chr_pos: 15_79531511 2
Duplicate SNP chr_pos: 9_45729853 2
Duplicate SNP chr_pos: 25_10359385 2
Duplicate SNP chr_pos: 22_56526462 2
Duplicate SNP chr_pos: 20_676757 2
Duplicate SNP chr_pos: 22_8308367 2
Duplicate SNP chr_pos: 20_17837675 2
Duplicate SNP chr_pos: 14_56761589 2
Duplicate SNP chr_pos: 3_76797698 2
Duplicate SNP chr_pos: 25_26191380 2
Duplicate SNP chr_pos: 19_43264699 2
Duplicate SNP chr_pos: 21_65198296 2
Duplicate SNP chr_pos: 8_106174871 2
Duplicate SNP chr_pos: 15_15367990 2
Duplicate SNP chr_pos: 2_111155237 2
Duplicate SNP chr_pos: 14_76524093 2
Duplicate SNP chr_pos: 13_71212300 2
Duplicate SNP chr_pos: 14_25401722 2
Duplicate SNP chr_pos: 12_80629629 2
Duplicate SNP chr_pos: 10_49791638 2
Duplicate SNP chr_pos: 6_54524018 2
Duplicate SNP chr_pos: 3_40654331 2
Duplicate SNP chr_pos: 15_15595454 2
Duplicate SNP chr_pos: 7_21298998 2
Duplicate SNP chr_pos: 15_15738972 2
Duplicate SNP chr_pos: 7_18454636 2
Duplicate SNP chr_pos: 7_80042191 2
Duplicate SNP chr_pos: 26_38233337 2
Duplicate SNP chr_pos: 28_35331560 2
Duplicate SNP chr_pos: 15_77675440 2
Duplicate SNP chr_pos: 15_15908105 2
Duplicate SNP chr_pos: 26_8221270 2
Duplicate SNP chr_pos: 18_1839733 2
Duplicate SNP chr_pos: 15_15697628 2
Duplicate SNP chr_pos: 11_1703612 2
Duplicate SNP chr_pos: 15_21207529 2
Duplicate SNP chr_pos: 15_79187295 2
Duplicate SNP chr_pos: 22_31841994 2
Duplicate SNP chr_pos: 20_51449833 2
Duplicate SNP chr_pos: 15_15544958 2
Duplicate SNP chr_pos: 15_15520367 2
Duplicate SNP chr_pos: 15_15656347 2
Duplicate SNP chr_pos: 15_15305071 2
Duplicate SNP chr_pos: 6_61079420 2
Duplicate SNP chr_pos: 15_15213319 2
Duplicate SNP chr_pos: 16_38116988 2
Duplicate SNP chr_pos: 28_44261945 2
Duplicate SNP chr_pos: 9_10783460 2
Duplicate SNP chr_pos: 14_48380429 2
Duplicate SNP chr_pos: 29_13695183 2
Duplicate SNP chr_pos: 1_136603695 2
Duplicate SNP chr_pos: 15_15162470 2
Duplicate SNP chr_pos: 15_15245842 2
Duplicate SNP chr_pos: 3_116448759 2
Duplicate SNP chr_pos: 15_38078775 2
Duplicate SNP chr_pos: 15_15190242 2
Duplicate SNP chr_pos: 8_95410507 2
Duplicate SNP chr_pos: 3_91910014 2
Duplicate SNP chr_pos: 17_60069612 2
Duplicate SNP chr_pos: 9_98483346 2
Duplicate SNP chr_pos: 12_79289169 2
Duplicate SNP chr_pos: 1_151349514 2
Duplicate SNP chr_pos: 4_17200594 2
Duplicate SNP chr_pos: 19_47734925 2
Duplicate SNP chr_pos: 26_47781927 2
Duplicate SNP chr_pos: 8_88974063 2
Duplicate SNP chr_pos: 14_29987025 2
Duplicate SNP chr_pos: 14_31322421 2
Duplicate SNP chr_pos: 14_21343649 2
Duplicate SNP chr_pos: 1_1277227 2
Duplicate SNP chr_pos: 24_4118163 2
Duplicate SNP chr_pos: 13_49053256 2
Duplicate SNP chr_pos: 10_55611885 2
Duplicate SNP chr_pos: 28_8508619 2
Duplicate SNP chr_pos: 6_55756520 2
Duplicate SNP chr_pos: 15_15949175 2
Duplicate SNP chr_pos: 4_94176209 2
Duplicate SNP chr_pos: 14_23893220 2
Duplicate SNP chr_pos: 25_26198573 2
Duplicate SNP chr_pos: 16_33671664 2
Duplicate SNP chr_pos: 3_71860062 2
Duplicate SNP chr_pos: 5_63150400 2
Duplicate SNP chr_pos: 7_37537822 2
Duplicate SNP chr_pos: 9_32549297 2
93개의 염색체 번호와 위치에서 염색체 번호와 위치는 같은데 서로 다른 SNP 이름을 갖고 있다는 에러를 뿌려줌. 유전체 선발에서는 제외를 하겠지만 여기서는 제외를 해야 올바른 것인지 잘 모르겠다. 분석에서 93쌍 즉 186개 SNP를 이용했는지 안 했는지 모르겠다.
Pair parent/progeny tested: 1403
Pair with conflicts: 19 1.4 %
1403개 부모/자식을 테스트해서 19개 관계가 친자감정 불일치
Sire-progeny tested: 715
Sire-progeny with conflicts: 11 1.5 %
715개 아비/자식을 테스트해서 11개 관계가 불일치
Dam-progeny tested: 688
Dam-progeny with conflicts: 8 1.2 %
688개 어미/자식을 테스트해서 8개 관계가 불일치
parent_progeny_conflicts.txt
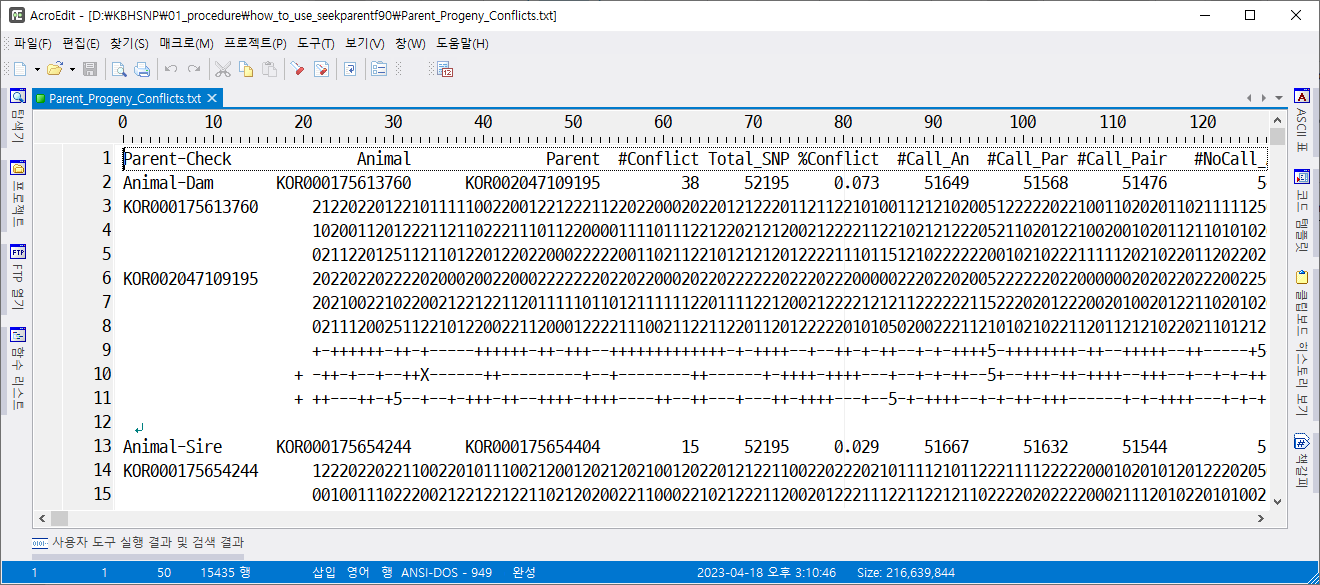
개체와 부모의 친자감정 결과를 보여준다. 그러나 너무 자세해서 보기 어렵다.
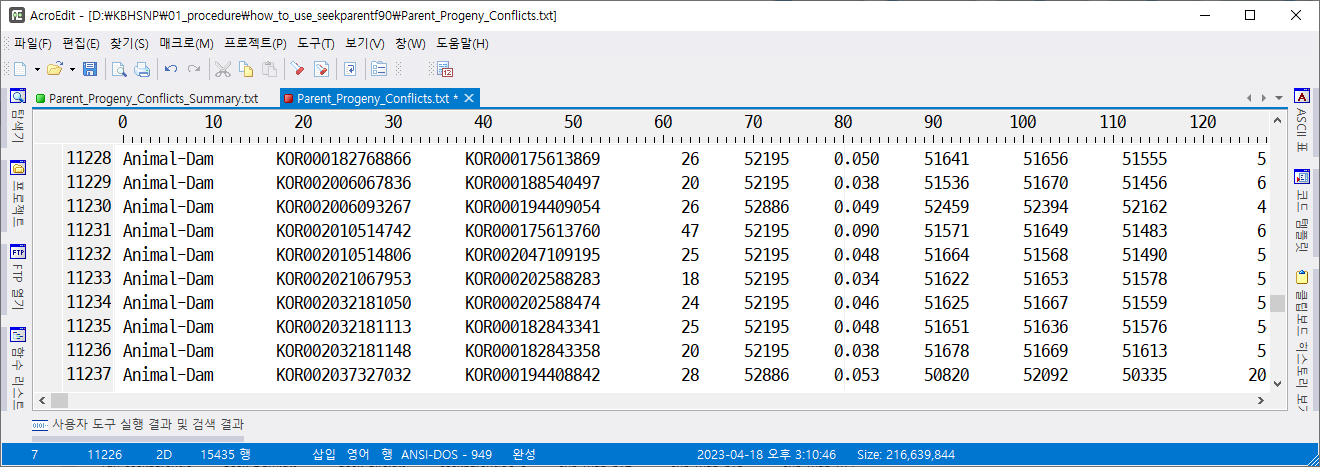
정렬을 해서 animal-dam 그리고 animal-sire 부분을 엑셀로 복사하고, D열을 내림차순 정렬하면 친자감정 불일치 개체 19두를 찾을 수 있다.
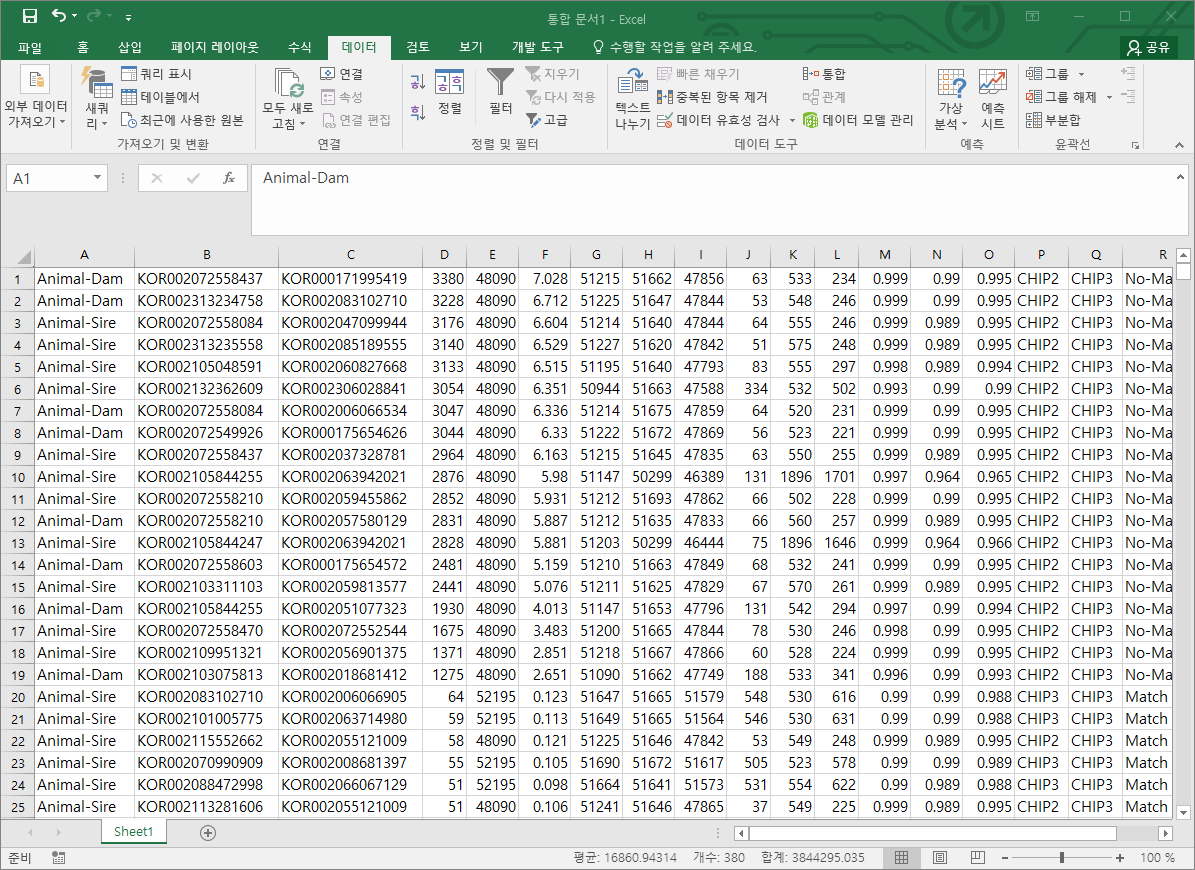
친자감정 불일치가 일어나는 경우는 세 가지이다. 1) 부(모)의 유전자형이 잘못 되었다. 2) 자손의 유전자형이 잘못되었다. 3) 혈통이 잘못되었다.
친자감정 불일치가 일어날 경우 1)과 2)를 생각안하고 무턱대고 3)을 생각할 경우 심각한 문제를 초래할 수 있다. 그러면 유전자형이 정말 제대로 되었는지는 어떻게 알 수 있는가?
parent_progeny_conflicts_summary.txt를 보자.
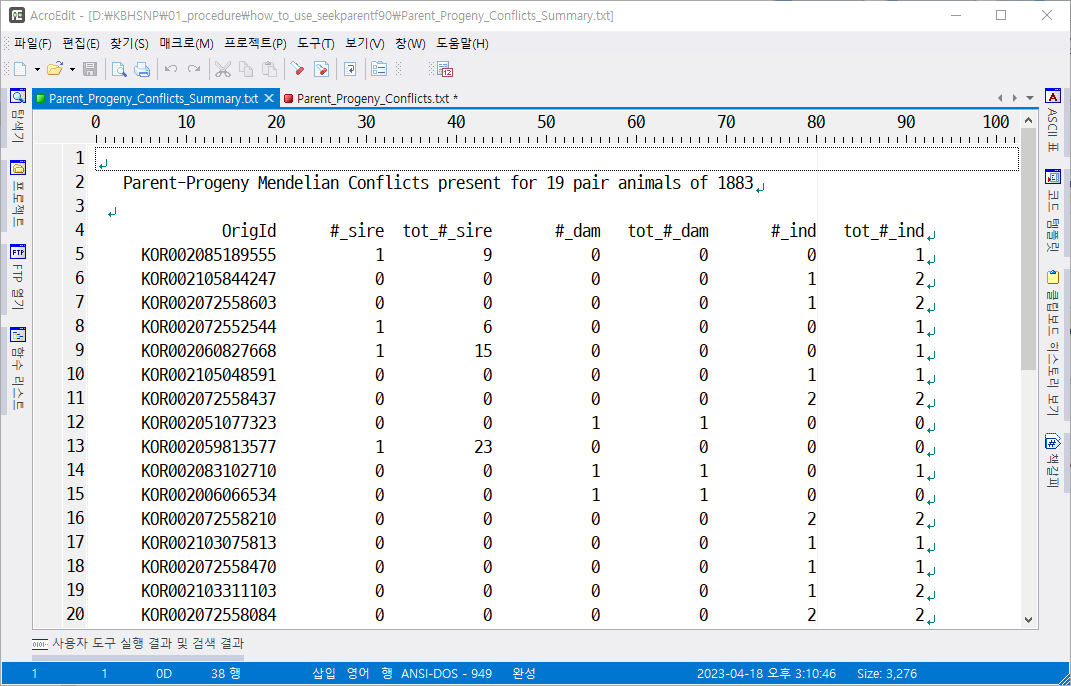
KOR002085189555 개체의 경우는 아비로서 9번 친자감정을 했고 그중 1번만 불일치다. 그러면 8번은 맞은 경우이므로 자기 자신의 유전자형이라고 확신할 수 있다.
그러나 KOR002072558437 는 개체로서 2번 친자감정 되었는데 2번다 친자감정 불일치가 났다. 이런 경우 자신의 유전자형이라고 보기 매우 힘들다. (아니라는 확신은 못한다.) 다시 genotyping을 할 수 있다면 하고, 다른 친자감정 결과 예를 들어 Microsatellite 친자감정 결과가 있다면 참고하여 결정하여야 한다.
조직을 샘플링할 때, 실험할 때, 결과를 처리할 때 등등 자신의 유전자형이 아닐 확률은 너무나 많다. 위와 같이 친자감정 일치가 하나라도 나왔다면 자신의 유전자형이라고 확신할 수 있지만 친자감정 일치가 없다면 자신의 유전자형인지 강한 의심을 하고, 그다음으로 혈통을 고민해야 한다.
A라는 개체가 B의 유전자형을 들고 우수한 개체로 판명이 나고, 그 결과를 이용하여 부모로 자손을 생산한다면 얼마나 큰 실수인가.
아래는 자신의 유전자형을 확신할 때 보는 부분이다. 즉 자신의 유전자형이 맞으므로 혈통을 수정해야 할 경우 보는 부분이다.
Seek method: Only for animals with conflicts
Total number of animals: 11
Found: 7
Not Found: 4
Found but cannot assign: 0
아비 불일치 11개 중 7개를 찾았다.
Seek method: Only for animals with conflicts
Total number of animals: 8
Found: 3
Not Found: 5
어미 불일치 8개 중 3개를 찾고, 5개는 찾지 못했다.
Output files
Pedigree file with removed parents for animals with conflicts: "Check_kbh_pedigree_yob.txt“
불일치인 개체의 부 또는 모 정보를 제거한 혈통 파일
아비 찾기 결과 seek_sire.txt
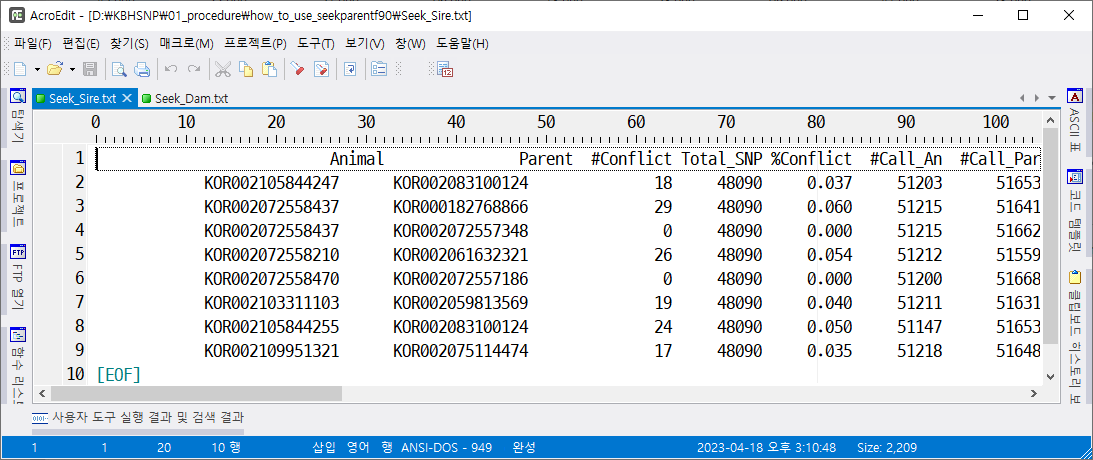
위에선 7개의 아비를 찾았다고 했으나 8개를 찾은 결과가 나옴. 이건 뭐지?
seek_dam.txt

3개의 어미를 찾았다.
이렇게 자기 자신의 올바른 유전자형과 올바른 혈통이 있다면 imputation 정확도가 매우 높아진다.
요약
1) 파일 준비
* genotype file
- kbh_bv2.txt
- kbh_bv3.txt
- kbh_hv1.txt
* chip 번호 넣는 awk 파일과 awk을 실행시키는 batch 파일
- insert_chip_number_bv2.awk, run_insert_chip_number_bv2.awk.bat
- insert_chip_number_bv3.awk, run_insert_chip_number_bv3.awk.bat
- insert_chip_number_hv1.awk, run_insert_chip_number_hv1.awk.bat
* chip 번호 넣은 genotype file을 합치는 파일
- make_one_genotype_file.bat
* map file
- snp_map_bv2
- snp_map_bv3
- snp_map_hv1
* 통합 map file을 만드는 SAS 프로그램
- make_snpmapfile_for_seekparentf90.sas
* 혈통 파일
- kbh_pedigree_yob.txt
* seekparentf90 실행 파일
- run_seekparentf90.bat
2) 실행
* genotyp file 준비
- run_insert_chip_number_bv2.awk.bat
- run_insert_chip_number_bv3.awk.bat
- run_insert_chip_number_hv1.awk.bat
- make_one_genotype_file.bat
* map file 준비
- make_snpmapfile_for_seekparentf90.sas (새로운 칩이 추가 되지 않는다면 기존 결과(mapfile_for_seekparentf90.txt) 사용 가능)
* seekparentf90 실행
- run_seekparentf90.bat
'Animal Breeding > Genomic Selection' 카테고리의 다른 글
| pregsf90으로 유전체 근교 계수 구하기 (0) | 2023.09.14 |
|---|---|
| H 행렬, H 행렬의 역행렬 유도 (0) | 2021.01.19 |
| SNP Effect를 이용한 DGV(or GEBV) 추정 (0) | 2020.03.05 |
| PostGSF90을 이용한 GWAS (0) | 2020.03.05 |
| SNP Marker Effect 추정 (0) | 2020.03.02 |