Bovine의 경우 여러 종류의 BeadChip이 있다. BovineSNP50 v2와 v3가 있을 뿐만 아니라 한국에서는 HanwooSNP50 v1을 사용하고 있다. 이것을 같이 분석하려면 한 쪽 버전으로 바꾸어야 하는데 이때 없는 유전자형을 추정해야 하는데 이것을 imputation이라고 한다.
예를 들어 각 BeadChip에 다음과 같은 SNP가 있다고 하자.
| SNP Name | BovineSNP50 v3 | HanwooSNP50 v1 |
| SNP1 | v | |
| SNP2 | v | v |
| SNP3 | v | |
| SNP4 | v | v |
| SNP5 | v | |
| SNP6 | v | v |
| SNP7 | v | |
| SNP8 | v | v |
| SNP9 | v | v |
| SNP10 | v |
BovineSNP50 v3를 기준으로 분석을 한다면 HanwooSNP50 v1에는 없는 SNP1, SNP5의 유전자형을 imputation해야 한다. 그리고 BovineSNP50 v3에는 없는 SNP3, SNP7, SNP7 등은 분석에 사용하지 않는다. 즉 HanwooSNP50 v1에 있는 SNP3, SNP7, SNP7 유전자형은 버린다.
imputation에 사용할 수 있는 다양한 도구가 있는데 여기서는 FImpute 프로그램을 이용한다.
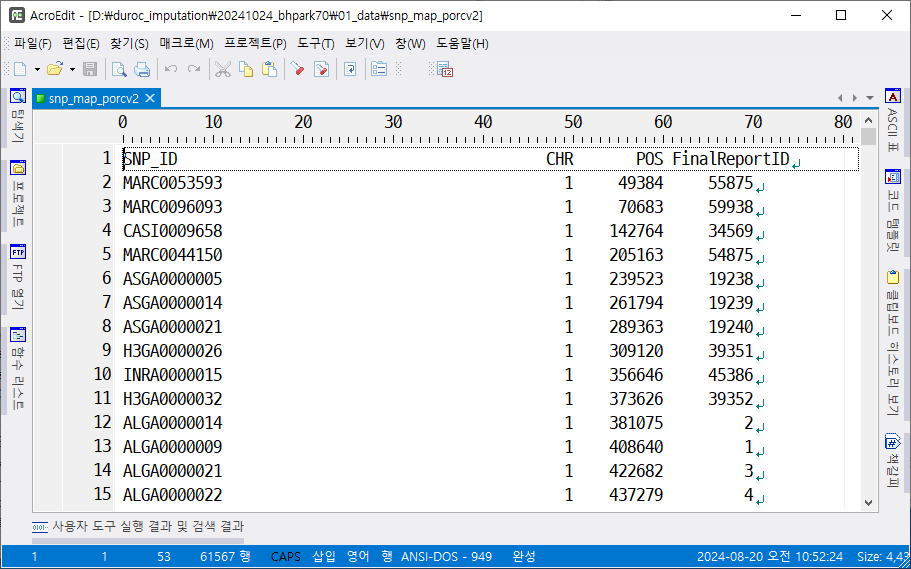
이전에 다운로드 받은 FinalReport의 유전자형을 변환했는데 그 결과는 다음과 같았다.

유전자형이 나와 있는데 이게 무슨 SNP인지는 snp_map에 있었다.

snp_map 파일을 통해서 몇 번째가 무슨 SNP인지 알 수 있는 것이다. 두 개 버전의 BeadChip을 이용하므로 어느 버전의 SNP는 사용하고, 어느 버전의 SNP는 사용하지 않을 것이라는 것을 알려주는 파일(snp_info 파일)을 작성해야 한다. FImpute에서 제공하는 예제에는 다음과 같이 나와 있다.

파일을 보면 염색체, SNP 위치 순으로 SNP를 정렬하고 chip1에 있고 없는 SNP, chip2에 있고 없는 SNP를 표시한 것을 알 수 있다.
그리고 어느 개체는 chip1이고 어느 개체는 chip2인지는 다음과 같이 표시한다.

개체 ID와 SNP 유전자형 사이에 1, 2와 같은 chip 번호가 들어가 있는 것을 알 수 있다.
FImpute 프로그램을 이용하여 imputation을 하기 위해서 할 일은 유전자형 파일에 chip 번호를 넣고, snp_info 파일을 만들고, 프로그램을 실행할 때 필요한 control 파일(snp_info 파일, 유전자형 파일, 혈통 파일의 위치와 이름을 알려주고, imputation 옵션을 설정하는 파일)을 작성하는 것이다.
여기서는 BovineSNP50v3을 HanwooSNP50v1로 imputation하는 것을 설명한다. 이전 설명을 참고하여 유전자형 변환을 완료한 것으로 본다. 준비한 파일은 다음과 같다.

BovineSNP50v3 유전자형 변환 파일

BovineSNP50v3 SNP Map 파일

HanwooSNP50v1 유전자형 변환 파일

HanwooSNP50v1 SNP Map 파일
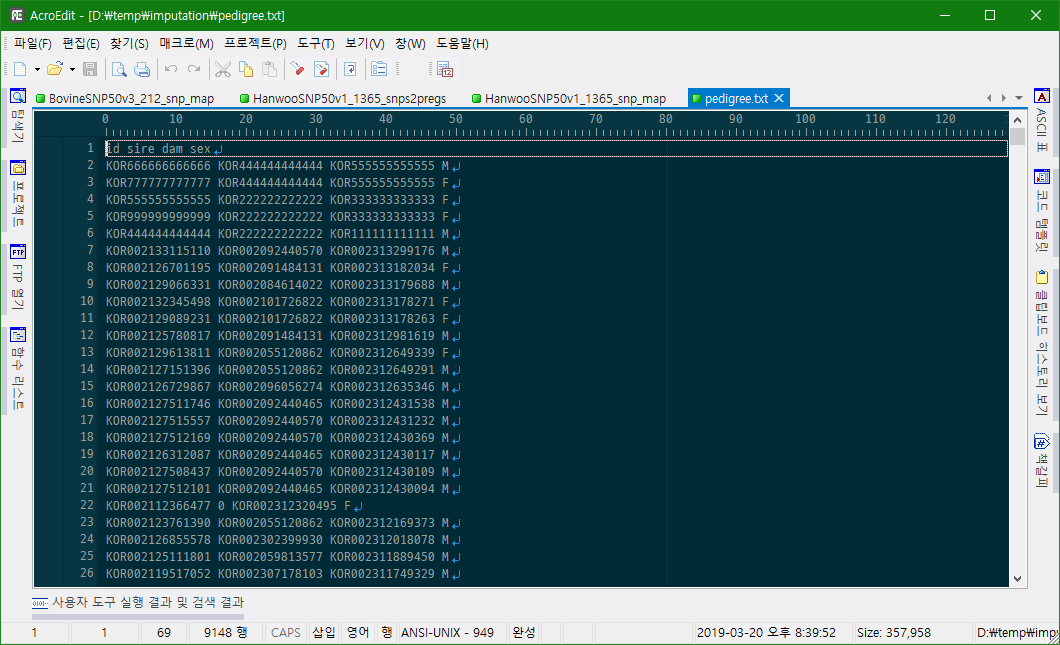
성별이 표시된 혈통 파일
■ 칩번호 넣기
위의 파일을 이용하여 HanwooSNP50v1이 reference popluation이므로 다음과 같은 파일을 만들어야 한다.
| ID Chip Call... 123456789 1 02202020202200222011120200020020 ...... 123456790 1 02101011102200222000020200120020 ...... |
다음과 같은 awk 프로그램을 이용한다.
insert_chip_number_HanwooSNP50v1.awk
| BEGIN{ print "ID Chip Call..." } { print $1 " 1 " $2 > "HanwooSNP50v1_for_fimpute_01.txt" } END { } |
먼저 1행에 “ID Chip Call...”을 출력하고, 이어서 입력파일의 1열과 2열을 읽어 출력하는데 두 열 가운데 “ 1 ” 삽입하고 HanwooSNP50v1_for_fimpute_01.txt로 출력한다는 의미이다. 실행을 편리하게하기 위하여 다음과 같이 배치 파일을 만든다.
run_insert_chip_number_HanwooSNP50v1.awk.bat
| awk -f insert_chip_number_HanwooSNP50v1.awk HanwooSNP50v1_1365_snps2pregs pause |
실행하려는 awk 프로그램 이름과 입력파일(HanwooSNP50v1_1365_snps2pregs)이다. 탐색기에서 더블 클릭하여 실행할 수 있다.
실행 화면

실행한 결과는 출력된 HanwooSNP50v1_for_fimpute_01.txt은 다음과 같다.
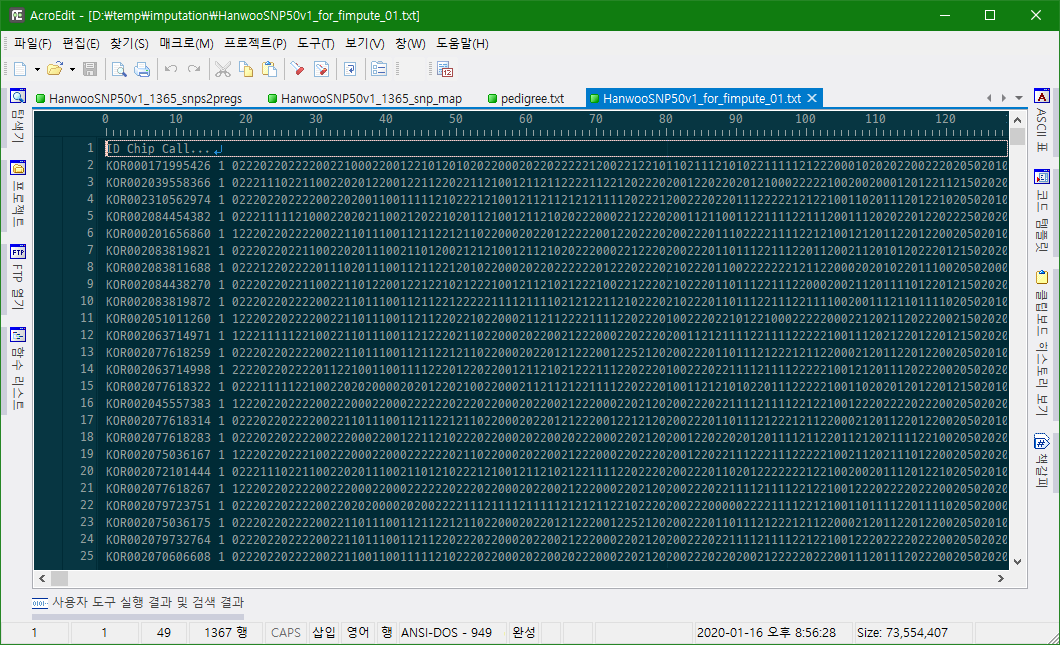
칩 번호 1(reference population)이 들어가 있는 것을 볼 수 있다.
위와 같은 방식으로 준비한 BovineSNP50v3의 genotype file은 아래와 같다.

■ snp_info 파일 만들기
snp_info.txt 파일의 형태는 위에서 본 바와 같다.
SNP_ID는 SNP의 이름, Chr은 염색체 번호, Pos는 염색체위의 SNP의 위치, Chip1은 첫째 칩에서의 SNP의 순서, Chip2는 둘째 칩에서의 SNP의 순서. chip1이 reference population, chip2가 target population.
Imputation을 하기 전에 각 BeadChip에 들어 있는 SNP를 파악할 필요가 있다. 다음은 각 BeadChip에 있는 SNP들의 염색체별 분포이다.
| Chromosome | BovineSNP50v3 | HanwooSNP50v1 |
| 1 | 3,273 | 3,225 |
| 2 | 2,671 | 2,756 |
| 3 | 2,455 | 2,582 |
| 4 | 2,409 | 2,479 |
| 5 | 2,177 | 2,156 |
| 6 | 2,885 | 3,158 |
| 7 | 2,518 | 2,481 |
| 8 | 2,326 | 2,246 |
| 9 | 2,124 | 2,077 |
| 10 | 2,302 | 2,357 |
| 11 | 2,163 | 2,181 |
| 12 | 1,688 | 1,651 |
| 13 | 1,700 | 1,684 |
| 14 | 1,689 | 2,274 |
| 15 | 1,668 | 1,681 |
| 16 | 1,643 | 1,599 |
| 17 | 1,560 | 1,567 |
| 18 | 1,308 | 1,303 |
| 19 | 1,367 | 1,380 |
| 20 | 1,587 | 1,571 |
| 21 | 1,425 | 1,398 |
| 22 | 1,214 | 1,212 |
| 23 | 1,106 | 1,124 |
| 24 | 1,223 | 1,230 |
| 25 | 915 | 939 |
| 26 | 1,035 | 1,032 |
| 27 | 934 | 918 |
| 28 | 901 | 905 |
| 29 | 1,012 | 1,029 |
| 30 | 1,157 | 947 |
| 31 | 11 | 11 |
| 32 | 759 | 700 |
| 33 | 13 | 13 |
| 소계 | 53,218 | 53,866 |
30번 염색체는 X 염색체를, 31번 염색체는 Y 염색체를, 32번 염색체는 MT를, 33번 염색체는 염색체 번호를 모르는 염색체를 의미한다.
Imputation을 하기 위해서는 SNP가 어느 염색체, 어느 위치에 있는지 알아야 하므로, Bovine의 경우 1에서 29번 염색체의 SNP만 사용한다.
즉 BovineSNP50v3에서는 51,278개의 SNP만을, HanwooSNP50v1에서는 52,195개의 SNP만을 이용한다.
염색체별로 BovineSNP50v3에만, 공통으로, HanwooSNP50v1에만 있는 SNP 개수는 다음과 같다.
| Chromosome | BovineSNP50v3 | BovineSNP50v3 + HanwooSNP50v1 | HanwooSNP50v1 | 소계 |
| 1 | 208 | 3,065 | 160 | 3,433 |
| 2 | 155 | 2,516 | 240 | 2,911 |
| 3 | 125 | 2,330 | 252 | 2,707 |
| 4 | 171 | 2,238 | 241 | 2,650 |
| 5 | 156 | 2,021 | 135 | 2,312 |
| 6 | 140 | 2,745 | 413 | 3,298 |
| 7 | 146 | 2,372 | 109 | 2,627 |
| 8 | 182 | 2,144 | 102 | 2,428 |
| 9 | 184 | 1,940 | 137 | 2,261 |
| 10 | 142 | 2,160 | 197 | 2,499 |
| 11 | 159 | 2,004 | 177 | 2,340 |
| 12 | 113 | 1,575 | 76 | 1,764 |
| 13 | 105 | 1,595 | 89 | 1,789 |
| 14 | 84 | 1,605 | 669 | 2,358 |
| 15 | 94 | 1,574 | 107 | 1,775 |
| 16 | 129 | 1,514 | 85 | 1,728 |
| 17 | 85 | 1,475 | 92 | 1,652 |
| 18 | 97 | 1,211 | 92 | 1,400 |
| 19 | 74 | 1,293 | 87 | 1,454 |
| 20 | 71 | 1,516 | 55 | 1,642 |
| 21 | 88 | 1,337 | 61 | 1,486 |
| 22 | 76 | 1,138 | 74 | 1,288 |
| 23 | 47 | 1,059 | 65 | 1,171 |
| 24 | 76 | 1,147 | 83 | 1,306 |
| 25 | 59 | 856 | 83 | 998 |
| 26 | 72 | 963 | 69 | 1,104 |
| 27 | 52 | 882 | 36 | 970 |
| 28 | 43 | 858 | 47 | 948 |
| 29 | 55 | 957 | 72 | 1,084 |
| 30 | 211 | 946 | 1 | 1,158 |
| 31 | - | 11 | - | 11 |
| 32 | 59 | 700 | - | 759 |
| 33 | - | 13 | - | 13 |
| 소계 | 3,458 | 49,760 | 4,106 | 57,324 |
그런데 염색체 번호와 위치가 같은 SNP들이 BovineSNP50v3에는 29쌍(SNP 58개)이 있고, HanwooSNPv1에는 73쌍(SNP 146개)이 있다. 이들을 삭제하여야 한다. 자세한 리스트는 다음과 같다.
BovineSNP50v2에서 염색체, 위치 중복인 SNP
| 번호 | Name | Chromosome | Position |
| 1 | UA-IFASA-2167 | 1 | 59409838 |
| 2 | ARS-USMARC-Parent-DQ404150-rs29012530 | 1 | 59409838 |
| 3 | Hapmap35832-SCAFFOLD197372_885 | 1 | 151349514 |
| 4 | ARS-USMARC-Parent-DQ404151-rs29019282 | 1 | 151349514 |
| 5 | Hapmap36382-SCAFFOLD210095_19074 | 2 | 111155237 |
| 6 | ARS-USMARC-Parent-DQ786757-rs29019900 | 2 | 111155237 |
| 7 | Hapmap52375-rs29010802 | 3 | 58040470 |
| 8 | ARS-USMARC-Parent-DQ435443-rs29010802 | 3 | 58040470 |
| 9 | Hapmap38870-BTA-01737 | 3 | 116448759 |
| 10 | ARS-USMARC-Parent-DQ839235-rs29012691 | 3 | 116448759 |
| 11 | Hapmap58054-rs29014143 | 4 | 17200594 |
| 12 | ARS-USMARC-Parent-DQ647186-rs29014143 | 4 | 17200594 |
| 13 | Hapmap33892-BES6_Contig314_677 | 4 | 94176209 |
| 14 | ARS-USMARC-Parent-DQ485413-no-rs | 4 | 94176209 |
| 15 | Hapmap36218-SCAFFOLD41765_2717 | 7 | 18454636 |
| 16 | ARS-USMARC-Parent-DQ786758-rs29024430 | 7 | 18454636 |
| 17 | UA-IFASA-2827 | 8 | 88974063 |
| 18 | ARS-USMARC-Parent-DQ837644-rs29010468 | 8 | 88974063 |
| 19 | Hapmap36391-SCAFFOLD165033_11046 | 8 | 106174871 |
| 20 | ARS-USMARC-Parent-DQ674265-rs29011266 | 8 | 106174871 |
| 21 | UA-IFASA-1922 | 9 | 45729853 |
| 22 | ARS-USMARC-Parent-DQ846689-rs29011985 | 9 | 45729853 |
| 23 | UA-IFASA-2515 | 9 | 98483346 |
| 24 | ARS-USMARC-Parent-DQ786765-rs29009858 | 9 | 98483346 |
| 25 | Hapmap59786-rs29012019 | 10 | 55611885 |
| 26 | ARS-USMARC-Parent-DQ984827-rs29012019 | 10 | 55611885 |
| 27 | Hapmap57799-rs29012894 | 11 | 1703612 |
| 28 | ARS-USMARC-Parent-DQ837646-rs29012894 | 11 | 1703612 |
| 29 | Hapmap36566-SCAFFOLD135238_3808 | 12 | 80629629 |
| 30 | ARS-USMARC-Parent-DQ832700-rs29012872 | 12 | 80629629 |
| 31 | Hapmap36096-SCAFFOLD140080_30362 | 13 | 25606469 |
| 32 | ARS-USMARC-Parent-EF034081-rs29009668 | 13 | 25606469 |
| 33 | Hapmap35881-SCAFFOLD20653_10639 | 14 | 48380429 |
| 34 | ARS-USMARC-Parent-DQ846691-rs29019814 | 14 | 48380429 |
| 35 | Hapmap35077-BES9_Contig405_919 | 15 | 21207529 |
| 36 | ARS-USMARC-Parent-EF042090-no-rs | 15 | 21207529 |
| 37 | Hapmap34596-BES7_Contig444_1293 | 15 | 38078775 |
| 38 | ARS-USMARC-Parent-DQ866817-no-rs | 15 | 38078775 |
| 39 | UA-IFASA-5162 | 15 | 79187295 |
| 40 | ARS-USMARC-Parent-DQ866818-rs29011701 | 15 | 79187295 |
| 41 | Hapmap57363-rs29014953 | 18 | 1839733 |
| 42 | ARS-USMARC-Parent-EF028073-rs29014953 | 18 | 1839733 |
| 43 | Hapmap59181-rs29010004 | 20 | 676757 |
| 44 | ARS-USMARC-Parent-DQ984828-rs29010004 | 20 | 676757 |
| 45 | Hapmap34041-BES1_Contig298_838 | 20 | 17837675 |
| 46 | ARS-USMARC-Parent-DQ888313-no-rs | 20 | 17837675 |
| 47 | Hapmap35417-SCAFFOLD255533_15525 | 21 | 65198296 |
| 48 | ARS-USMARC-Parent-EF026085-rs29021607 | 21 | 65198296 |
| 49 | Hapmap55319-rs29013532 | 22 | 56526462 |
| 50 | ARS-USMARC-Parent-EF034082-rs29013532 | 22 | 56526462 |
| 51 | Hapmap53362-rs29013727 | 26 | 8221270 |
| 52 | ARS-USMARC-Parent-DQ990834-rs29013727 | 26 | 8221270 |
| 53 | Hapmap35000-BES9_Contig272_944 | 26 | 38233337 |
| 54 | ARS-USMARC-Parent-EF034086-no-rs | 26 | 38233337 |
| 55 | Hapmap36071-SCAFFOLD106623_11509 | 28 | 35331560 |
| 56 | ARS-USMARC-Parent-EF026086-rs29013660 | 28 | 35331560 |
| 57 | Hapmap36794-SCAFFOLD186736_5402 | 28 | 44261945 |
| 58 | ARS-USMARC-Parent-EF042091-rs29014974 | 28 | 44261945 |
| 59 | Hapmap36059-SCAFFOLD50303_4748 | 29 | 28647816 |
| 60 | ARS-USMARC-Parent-EF034080-rs29024749 | 29 | 28647816 |
BovineSNP50v3에서 염색체, 위치 중복인 SNP
| 번호 | Name | Chromosome | Position |
| 1 | ARS-USMARC-Parent-DQ404150-rs29012530 | 1 | 59409838 |
| 2 | UA-IFASA-2167 | 1 | 59409838 |
| 3 | Hapmap35832-SCAFFOLD197372_885 | 1 | 151349514 |
| 4 | ARS-USMARC-Parent-DQ404151-rs29019282 | 1 | 151349514 |
| 5 | Hapmap52375-rs29010802 | 3 | 58040470 |
| 6 | ARS-USMARC-Parent-DQ435443-rs29010802 | 3 | 58040470 |
| 7 | Hapmap58054-rs29014143 | 4 | 17200594 |
| 8 | ARS-USMARC-Parent-DQ647186-rs29014143 | 4 | 17200594 |
| 9 | Hapmap33892-BES6_Contig314_677 | 4 | 94176209 |
| 10 | ARS-USMARC-Parent-DQ485413-no-rs | 4 | 94176209 |
| 11 | UA-IFASA-2827 | 8 | 88974063 |
| 12 | ARS-USMARC-Parent-DQ837644-rs29010468 | 8 | 88974063 |
| 13 | Hapmap36391-SCAFFOLD165033_11046 | 8 | 106174871 |
| 14 | ARS-USMARC-Parent-DQ674265-rs29011266 | 8 | 106174871 |
| 15 | UA-IFASA-1922 | 9 | 45729853 |
| 16 | ARS-USMARC-Parent-DQ846689-rs29011985 | 9 | 45729853 |
| 17 | UA-IFASA-2515 | 9 | 98483346 |
| 18 | ARS-USMARC-Parent-DQ786765-rs29009858 | 9 | 98483346 |
| 19 | Hapmap59786-rs29012019 | 10 | 55611885 |
| 20 | ARS-USMARC-Parent-DQ984827-rs29012019 | 10 | 55611885 |
| 21 | Hapmap57799-rs29012894 | 11 | 1703612 |
| 22 | ARS-USMARC-Parent-DQ837646-rs29012894 | 11 | 1703612 |
| 23 | Hapmap36566-SCAFFOLD135238_3808 | 12 | 80629629 |
| 24 | ARS-USMARC-Parent-DQ832700-rs29012872 | 12 | 80629629 |
| 25 | Hapmap36096-SCAFFOLD140080_30362 | 13 | 25606469 |
| 26 | ARS-USMARC-Parent-EF034081-rs29009668 | 13 | 25606469 |
| 27 | Hapmap35881-SCAFFOLD20653_10639 | 14 | 48380429 |
| 28 | ARS-USMARC-Parent-DQ846691-rs29019814 | 14 | 48380429 |
| 29 | Hapmap35077-BES9_Contig405_919 | 15 | 21207529 |
| 30 | ARS-USMARC-Parent-EF042090-no-rs | 15 | 21207529 |
| 31 | Hapmap34596-BES7_Contig444_1293 | 15 | 38078775 |
| 32 | ARS-USMARC-Parent-DQ866817-no-rs | 15 | 38078775 |
| 33 | UA-IFASA-5162 | 15 | 79187295 |
| 34 | ARS-USMARC-Parent-DQ866818-rs29011701 | 15 | 79187295 |
| 35 | Hapmap57363-rs29014953 | 18 | 1839733 |
| 36 | ARS-USMARC-Parent-EF028073-rs29014953 | 18 | 1839733 |
| 37 | Hapmap59181-rs29010004 | 20 | 676757 |
| 38 | ARS-USMARC-Parent-DQ984828-rs29010004 | 20 | 676757 |
| 39 | Hapmap34041-BES1_Contig298_838 | 20 | 17837675 |
| 40 | ARS-USMARC-Parent-DQ888313-no-rs | 20 | 17837675 |
| 41 | Hapmap35417-SCAFFOLD255533_15525 | 21 | 65198296 |
| 42 | ARS-USMARC-Parent-EF026085-rs29021607 | 21 | 65198296 |
| 43 | LTF | 22 | 31841994 |
| 44 | Hapmap40441-BTA-54131 | 22 | 31841994 |
| 45 | Hapmap55319-rs29013532 | 22 | 56526462 |
| 46 | ARS-USMARC-Parent-EF034082-rs29013532 | 22 | 56526462 |
| 47 | Hapmap53362-rs29013727 | 26 | 8221270 |
| 48 | ARS-USMARC-Parent-DQ990834-rs29013727 | 26 | 8221270 |
| 49 | Hapmap35000-BES9_Contig272_944 | 26 | 38233337 |
| 50 | ARS-USMARC-Parent-EF034086-no-rs | 26 | 38233337 |
| 51 | LYST | 28 | 8508619 |
| 52 | CHS1 | 28 | 8508619 |
| 53 | Hapmap36071-SCAFFOLD106623_11509 | 28 | 35331560 |
| 54 | ARS-USMARC-Parent-EF026086-rs29013660 | 28 | 35331560 |
| 55 | Hapmap36794-SCAFFOLD186736_5402 | 28 | 44261945 |
| 56 | ARS-USMARC-Parent-EF042091-rs29014974 | 28 | 44261945 |
| 57 | Hapmap36059-SCAFFOLD50303_4748 | 29 | 28647816 |
| 58 | ARS-USMARC-Parent-EF034080-rs29024749 | 29 | 28647816 |
HanwooSNP50v1에서 염색체, 위치 중복인 SNP
| 번호 | Name | Chromosome | Position |
| 1 | GART | 1 | 1277227 |
| 2 | rs465495560 | 1 | 1277227 |
| 3 | ARS-USMARC-Parent-DQ404150-rs29012530 | 1 | 59409838 |
| 4 | UA-IFASA-2167 | 1 | 59409838 |
| 5 | ARS-BFGL-NGS-12806 | 1 | 136603695 |
| 6 | rs110340232 | 1 | 136603695 |
| 7 | ARS-USMARC-Parent-DQ404151-rs29019282 | 1 | 151349514 |
| 8 | Hapmap35832-SCAFFOLD197372_885 | 1 | 151349514 |
| 9 | ARS-USMARC-Parent-DQ435443-rs29010802 | 3 | 58040470 |
| 10 | Hapmap52375-rs29010802 | 3 | 58040470 |
| 11 | Hapmap27208-BTA-157501 | 3 | 71860062 |
| 12 | NIAS_PAT_00114 | 3 | 71860062 |
| 13 | BTB-01509081 | 3 | 76797698 |
| 14 | rs42624577 | 3 | 76797698 |
| 15 | ARS-USMARC-Parent-DQ647186-rs29014143 | 4 | 17200594 |
| 16 | Hapmap58054-rs29014143 | 4 | 17200594 |
| 17 | ARS-USMARC-Parent-DQ485413-no-rs | 4 | 94176209 |
| 18 | Hapmap33892-BES6_Contig314_677 | 4 | 94176209 |
| 19 | APAF1 | 5 | 63150400 |
| 20 | rs448942533 | 5 | 63150400 |
| 21 | BovineHD0600016794 | 6 | 61079420 |
| 22 | rs136396635 | 6 | 61079420 |
| 23 | BTB-00986847 | 7 | 1071389 |
| 24 | NIAS_PAT_00012 | 7 | 1071389 |
| 25 | ARS-BFGL-NGS-76601 | 7 | 21298998 |
| 26 | rs110095569 | 7 | 21298998 |
| 27 | BTB-01273492 | 7 | 80042191 |
| 28 | NIAS_PAT_00104 | 7 | 80042191 |
| 29 | ARS-USMARC-Parent-DQ837644-rs29010468 | 8 | 88974063 |
| 30 | UA-IFASA-2827 | 8 | 88974063 |
| 31 | rs456206907 | 8 | 95410507 |
| 32 | SMC2 | 8 | 95410507 |
| 33 | ARS-USMARC-Parent-DQ674265-rs29011266 | 8 | 106174871 |
| 34 | Hapmap36391-SCAFFOLD165033_11046 | 8 | 106174871 |
| 35 | BovineHD0900002683 | 9 | 10783460 |
| 36 | rs111016555 | 9 | 10783460 |
| 37 | BTB-00386288 | 9 | 32549297 |
| 38 | rs43593958 | 9 | 32549297 |
| 39 | ARS-USMARC-Parent-DQ846689-rs29011985 | 9 | 45729853 |
| 40 | UA-IFASA-1922 | 9 | 45729853 |
| 41 | ARS-USMARC-Parent-DQ786765-rs29009858 | 9 | 98483346 |
| 42 | UA-IFASA-2515 | 9 | 98483346 |
| 43 | BTB-00426704 | 10 | 49791638 |
| 44 | NIAS_PAT_00123 | 10 | 49791638 |
| 45 | ARS-USMARC-Parent-DQ984827-rs29012019 | 10 | 55611885 |
| 46 | Hapmap59786-rs29012019 | 10 | 55611885 |
| 47 | ARS-USMARC-Parent-DQ837646-rs29012894 | 11 | 1703612 |
| 48 | Hapmap57799-rs29012894 | 11 | 1703612 |
| 49 | Hapmap34629-BES7_Contig224_786 | 11 | 33265117 |
| 50 | rs43710094 | 11 | 33265117 |
| 51 | ARS-USMARC-Parent-DQ832700-rs29012872 | 12 | 80629629 |
| 52 | Hapmap36566-SCAFFOLD135238_3808 | 12 | 80629629 |
| 53 | ARS-USMARC-Parent-EF034081-rs29009668 | 13 | 25606469 |
| 54 | Hapmap36096-SCAFFOLD140080_30362 | 13 | 25606469 |
| 55 | BTB-01186797 | 13 | 49053256 |
| 56 | NIAS_PAT_00018 | 13 | 49053256 |
| 57 | ARS-BFGL-NGS-28234 | 14 | 21343649 |
| 58 | rs109517821 | 14 | 21343649 |
| 59 | ARS-BFGL-BAC-8052 | 14 | 23893220 |
| 60 | rs110061498 | 14 | 23893220 |
| 61 | Hapmap46735-BTA-86653 | 14 | 25401722 |
| 62 | rs41657755 | 14 | 25401722 |
| 63 | Hapmap27112-BTC-063342 | 14 | 27271835 |
| 64 | rs42893390 | 14 | 27271835 |
| 65 | ARS-BFGL-NGS-2627 | 14 | 29987025 |
| 66 | rs43002316 | 14 | 29987025 |
| 67 | Hapmap50960-BTA-43399 | 14 | 31322421 |
| 68 | rs41639002 | 14 | 31322421 |
| 69 | ARS-USMARC-Parent-DQ846691-rs29019814 | 14 | 48380429 |
| 70 | Hapmap35881-SCAFFOLD20653_10639 | 14 | 48380429 |
| 71 | Hapmap23726-BTC-051363 | 14 | 56761589 |
| 72 | rs43134497 | 14 | 56761589 |
| 73 | Hapmap59332-rs29016542 | 15 | 15162470 |
| 74 | rs29016542 | 15 | 15162470 |
| 75 | rs29012762 | 15 | 15190242 |
| 76 | UA-IFASA-2285 | 15 | 15190242 |
| 77 | Hapmap58058-rs29012133 | 15 | 15213319 |
| 78 | rs29012133 | 15 | 15213319 |
| 79 | ARS-BFGL-NGS-10627 | 15 | 15245842 |
| 80 | rs109677489 | 15 | 15245842 |
| 81 | ARS-BFGL-BAC-31586 | 15 | 15305071 |
| 82 | rs108999006 | 15 | 15305071 |
| 83 | ARS-BFGL-NGS-10723 | 15 | 15367990 |
| 84 | rs109495422 | 15 | 15367990 |
| 85 | Hapmap35322-BES8_Contig457_1759 | 15 | 15520367 |
| 86 | rs43706895 | 15 | 15520367 |
| 87 | ARS-BFGL-NGS-17747 | 15 | 15544958 |
| 88 | rs110050515 | 15 | 15544958 |
| 89 | ARS-BFGL-NGS-17568 | 15 | 15656347 |
| 90 | rs109772143 | 15 | 15656347 |
| 91 | Hapmap35448-SCAFFOLD52197_3147 | 15 | 15697628 |
| 92 | rs29024927 | 15 | 15697628 |
| 93 | ARS-BFGL-NGS-12483 | 15 | 15908105 |
| 94 | rs109546387 | 15 | 15908105 |
| 95 | ARS-BFGL-NGS-69055 | 15 | 15949175 |
| 96 | rs110107689 | 15 | 15949175 |
| 97 | ARS-USMARC-Parent-EF042090-no-rs | 15 | 21207529 |
| 98 | Hapmap35077-BES9_Contig405_919 | 15 | 21207529 |
| 99 | ARS-USMARC-Parent-DQ866817-no-rs | 15 | 38078775 |
| 100 | Hapmap34596-BES7_Contig444_1293 | 15 | 38078775 |
| 101 | rs109636878 | 15 | 77675440 |
| 102 | LRP4_3 | 15 | 77675440 |
| 103 | ARS-USMARC-Parent-DQ866818-rs29011701 | 15 | 79187295 |
| 104 | UA-IFASA-5162 | 15 | 79187295 |
| 105 | ARS-BFGL-NGS-112854 | 16 | 33671664 |
| 106 | rs43131245 | 16 | 33671664 |
| 107 | ARS-USMARC-Parent-EF028073-rs29014953 | 18 | 1839733 |
| 108 | Hapmap57363-rs29014953 | 18 | 1839733 |
| 109 | g.43264699G>A | 19 | 43264699 |
| 110 | NAGLU | 19 | 43264699 |
| 111 | rs466131011 | 19 | 47734925 |
| 112 | MRC2_1 | 19 | 47734925 |
| 113 | ARS-USMARC-Parent-DQ984828-rs29010004 | 20 | 676757 |
| 114 | Hapmap59181-rs29010004 | 20 | 676757 |
| 115 | ARS-USMARC-Parent-DQ888313-no-rs | 20 | 17837675 |
| 116 | Hapmap34041-BES1_Contig298_838 | 20 | 17837675 |
| 117 | BTB-00787104 | 20 | 51449833 |
| 118 | rs41948047 | 20 | 51449833 |
| 119 | ARS-USMARC-Parent-EF026085-rs29021607 | 21 | 65198296 |
| 120 | Hapmap35417-SCAFFOLD255533_15525 | 21 | 65198296 |
| 121 | ARS-BFGL-NGS-74728 | 22 | 8308367 |
| 122 | NIAS_PAT_00057 | 22 | 8308367 |
| 123 | Hapmap40441-BTA-54131 | 22 | 31841994 |
| 124 | LTF | 22 | 31841994 |
| 125 | ARS-USMARC-Parent-EF034082-rs29013532 | 22 | 56526462 |
| 126 | Hapmap55319-rs29013532 | 22 | 56526462 |
| 127 | ARS-BFGL-BAC-42839 | 24 | 4118163 |
| 128 | NIAS_PAT_00131 | 24 | 4118163 |
| 129 | ATP2A1_1 | 25 | 26191380 |
| 130 | g.26191380C>T | 25 | 26191380 |
| 131 | ATP2A1_4 | 25 | 26198573 |
| 132 | g.26198573G>A | 25 | 26198573 |
| 133 | ARS-USMARC-Parent-DQ990834-rs29013727 | 26 | 8221270 |
| 134 | Hapmap53362-rs29013727 | 26 | 8221270 |
| 135 | ARS-USMARC-Parent-EF034086-no-rs | 26 | 38233337 |
| 136 | Hapmap35000-BES9_Contig272_944 | 26 | 38233337 |
| 137 | CHS1 | 28 | 8508619 |
| 138 | LYST | 28 | 8508619 |
| 139 | ARS-USMARC-Parent-EF026086-rs29013660 | 28 | 35331560 |
| 140 | Hapmap36071-SCAFFOLD106623_11509 | 28 | 35331560 |
| 141 | ARS-USMARC-Parent-EF042091-rs29014974 | 28 | 44261945 |
| 142 | Hapmap36794-SCAFFOLD186736_5402 | 28 | 44261945 |
| 143 | BTB-01008169 | 29 | 13695183 |
| 144 | NIAS_PAT_00031 | 29 | 13695183 |
| 145 | ARS-USMARC-Parent-EF034080-rs29024749 | 29 | 28647816 |
| 146 | Hapmap36059-SCAFFOLD50303_4748 | 29 | 28647816 |
다음은 illumina2pregs가 만들어 낸 snp_map 파일을 이용하여 FImpute에 사용할 snp_info 파일을 만드는 SAS 프로그램이다.
| /* file name : make_snp_info_for_fimpute.sas FImpute 프로그램을 위한 snp_info.txt 파일 만들기 BovineSNP50v3를 HanwooSNP50v1으로 Imputation할 때 필요한 snp_info.txt 만들기 */ /* BovineSNP50v3 snp_map 파일 읽기 */ data a1; infile 'BovineSNP50v3_snp_map'; informat Bv3Index best32. ; informat Bv3chrmo best32. ; informat Bv3Pos best32. ; informat Name $37. ; informat Bv3OIndex best32. ; format Bv3Index best32. ; format Bv3chrmo best32. ; format Bv3Pos best32. ; format Name $37. ; format Bv3OIndex best32. ; input Bv3Index Bv3chrmo Bv3Pos Name $ Bv3OIndex ; run; /* HanwooSNP50v1 snp_map 파일 읽기 */ data b1; infile 'HanwooSNP50v1_snp_map'; informat Hv1Index best32. ; informat Hv1chrmo best32. ; informat Hv1Pos best32. ; informat Name $37. ; informat Hv1OIndex best32. ; format Hv1Index best32. ; format Hv1chrmo best32. ; format Hv1Pos best32. ; format Name $37. ; format Hv1OIndex best32. ; input Hv1Index Hv1chrmo Hv1Pos Name $ Hv1OIndex ; run; /* 염색체별 SNP 빈도수 */ proc freq data = a1; table Bv3chrmo; run; proc freq data = b1; table Hv1chrmo; run; /* bv3와 hv1 머지하기 위하여 정렬 */ proc sort data = a1; by name; run; proc sort data = b1; by name; run; /* bv3와 hv1 머지하기 */ data c1; merge a1 b1; by name; run; /* snp_info 파일 만들기 */ data c2; set c1; if Bv3index = . then Bv3index = 0; if Hv1index = . then Hv1index = 0; chr = Bv3chrmo; if Bv3chrmo = . then chr = Hv1chrmo; pos = Bv3pos; if Bv3pos = . then pos = Hv1pos; if Bv3index ~= 0 and Hv1index ~= 0 then Bv3_Hv1 = "Bv3_Hv1"; else if Bv3index = 0 and Hv1index ~= 0 then Bv3_Hv1 = "Hv1"; else if Bv3index ~= 0 and Hv1index = 0 then Bv3_Hv1 = "Bv3"; run; proc freq data = c2; table chr * Bv3_Hv1 / nocol nopercent; run; /* 염색체 위치 순으로 정렬 */ proc sort data = c2; by chr pos; run; data _null_; set c2; file "snp_info_for_fimpute.txt"; if _n_ = 1 then put "SNP_ID Chr Pos HV1Index BV3Index"; put name @41 chr @51 pos @61 hv1index @71 bv3index; run; /* 제외시킬 SNP 찾기 */ /* 염색체와 위치가 같은 SNP, hv1에 없는 SNP */ /* 30번 이상 염색체는 fimpute에서 제외 */ /* 염색체 29 이하 선택 */ data c3; set c2; if chr <= 29 then output; run; /* 염색체 위치 순으로 정렬 */ proc sort data = c3; by chr pos; run; /* 같은 염색체와 위치 추출 */ data e1; set c3; flag1 = 1; keep chr pos flag1; if lag(chr) = chr and lag(pos) = pos then output; run; /* 염색체와 위치가 같은 SNP 추출 */ data e1_r1; merge e1 c3; by chr pos; if flag1 = 1 then output; run; /* SNP 이름만 남기기 */ data e1_r2; set e1_r1; keep name; run; /* hv1에 없는 SNP 추출 */ data e2; set c3; if hv1index = 0 then output; run; /* SNP 이름만 남기기 */ data e2_r1; set e2; keep name; run; /* 제외할 SNP 합치기 */ data ef1; set e1_r2 e2_r1; run; /* 중복 SNP 삭제 */ proc sort data = ef1; by name; run; data ef2; set ef1; by name; if first.name then output; run; /* 출력 */ data _null_; set ef2; file "excluded_snp_for_fimpute.txt"; put name; run; |
프로그램 실행 순서는 다음과 같다.
○ BovineSNP50v3 snp_map 파일 읽기
○ HanwooSNP50v1 snp_map 파일 읽기
○ SNP Name을 기준으로 두 파일 머지하기
○ snp_info 파일 출력(snp_info_for_fimpute.txt)
○ 분석에서 제외할 SNP 추출 및 출력(excluded_snp_for_fimpute.txt)
- 1번에서 29번 염새체 중
- 염색체 번호와 위치가 같은 SNP
- reference popuation을 위한 chip에 없는 SNP를 삭제
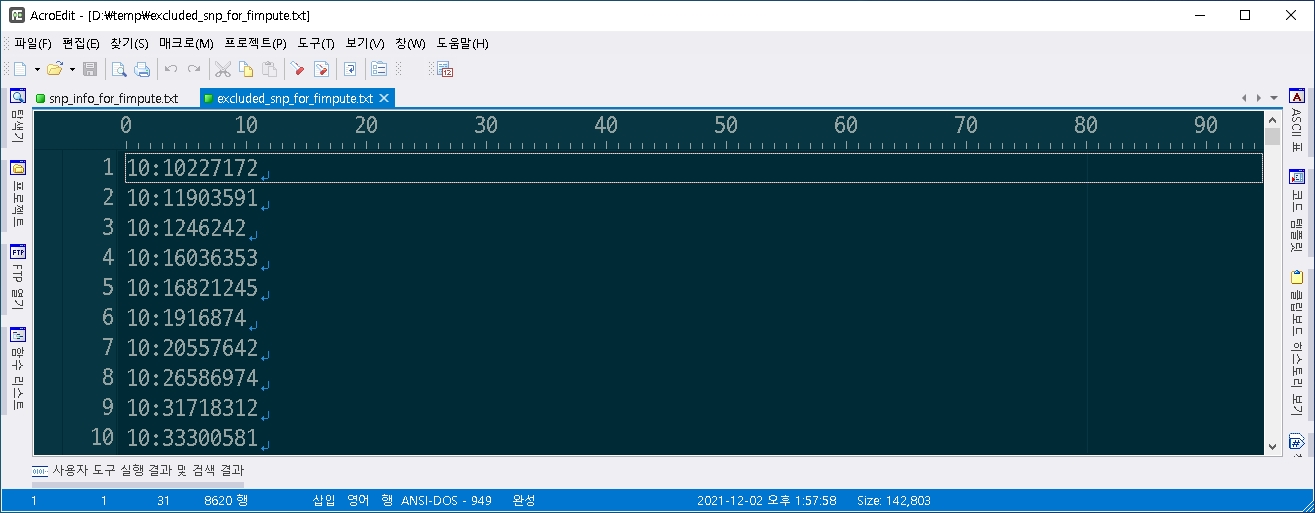
결과적으로 만들어진 snp_info_for_fimpute.txt는 다음과 같다.
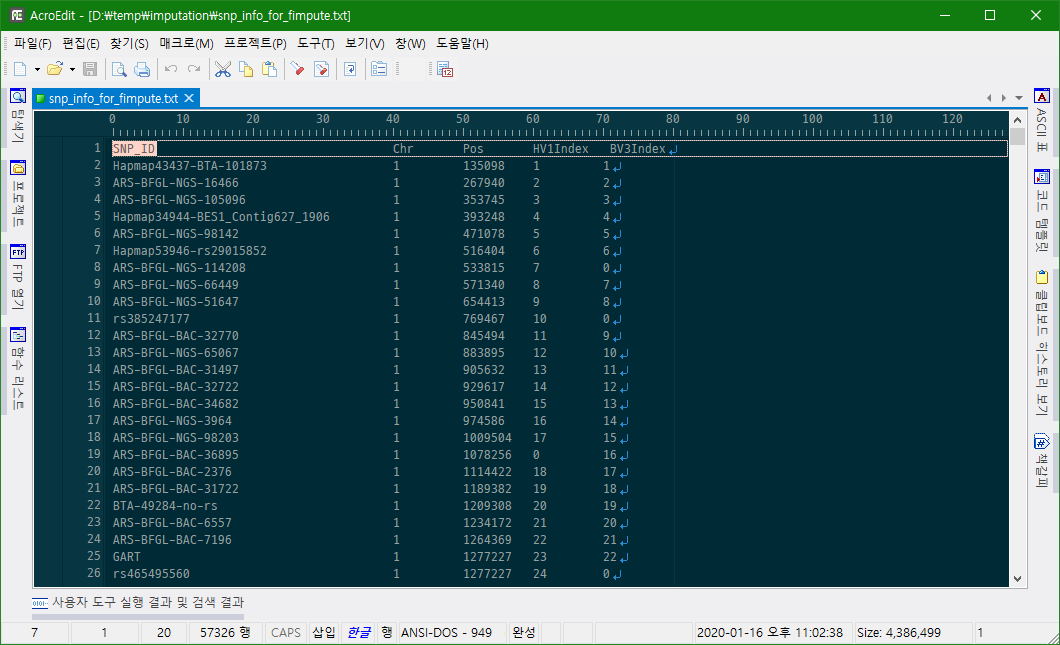
imputation에서 제외할 1 ~ 29번 염색체의 snp 이름을 담고 있는 excluded_snp_for_fimpute.txt는 다음과 같다. 1 ~ 29번 염색체의 3336개의 SNP를 제외할 것이다.

<2021.12.2.>
illumina2pregs가 만들어 내는 map file의 형식이 바뀌었다. 그래서 그걸 읽는 SAS 프로그램도 바뀌어야 한다. 여기서는 map file의 컬럼 순서가 snp이름, 염색체, 위치, 원래번호 등 이런 순으로 되어 있을 경우에 snp_info 파일을 만드는 경우를 설명한다. map file의 순서는 illumina2preGS 프로그램을 돌릴 때 --sortmap 옵션을 주어 염색체, 위치 순으로 정렬되어 있다고 가정한다.
bovine version 2의 map file
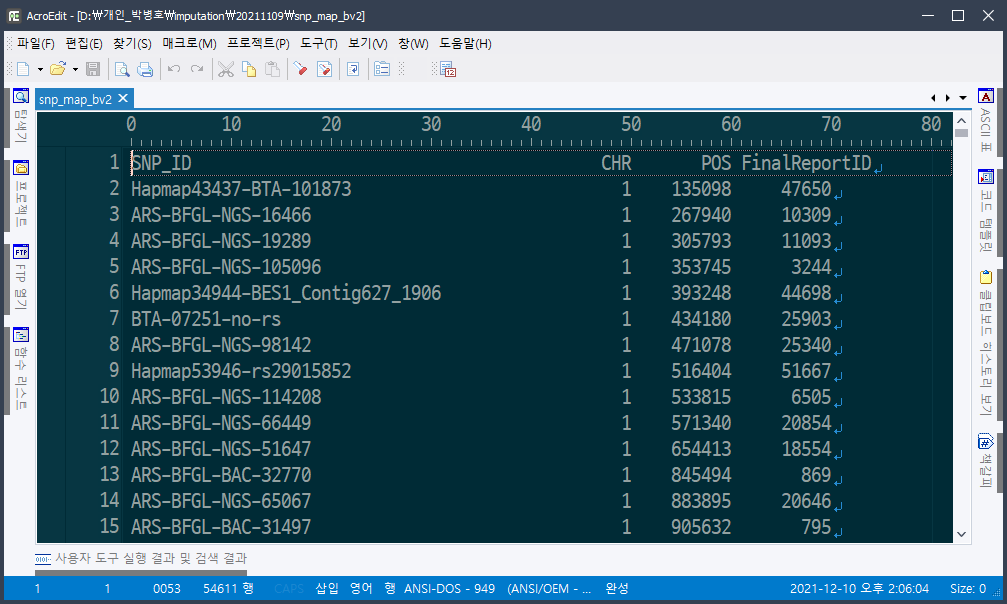
bovine version 3의 map file

hanwoo version 1의 map file

그래서 이번에는 bovine version2, bovine version 3, hanwoo version 1이 있을 때, illumina2preGS Version 1.3으로 변환하고 bovine version 3로 imputation할 경우 필요한 snp_info 파일을 만드는 sas 코드를 다시 만들었다. 전과 크게 다르지 않지만 chip이 세 개 이므로 그에 따른 작업이 조금 더 필요하다.
/*
file name : make_snp_info_for_fimpute.sas
FImpute 프로그램을 위한 snp_info.txt 파일 만들기
BovineSNP50v2, HanwooSNP50v1 -> BOvineSNP50v3로 Imputation할 때 필요한 snp_info.txt 만들기
*/
/* BovineSNP50v2 snp_map 파일 읽기 */
data a1 ;
infile 'snp_map_bv2' firstobs = 2;
informat name $37. ;
informat bv2chrmo best32. ;
informat bv2pos best32. ;
informat bv2oindex best32. ;
format name $37. ;
format bv2chrmo best32. ;
format bv2pos best32. ;
format bv2oindex best32. ;
input
name
bv2chrmo
bv2pos
bv2oindex
;
bv2index = _n_;
run;
/* BovineSNP50v3 snp_map 파일 읽기 */
data b1 ;
infile 'snp_map_bv3' firstobs = 2;
informat name $37. ;
informat bv3chrmo best32. ;
informat bv3pos best32. ;
informat bv3oindex best32. ;
format name $37. ;
format bv3chrmo best32. ;
format bv3pos best32. ;
format bv3oindex best32. ;
input
name
bv3chrmo
bv3pos
bv3oindex
;
bv3index = _n_;
run;
/* HanwooSNP50v1 snp_map 파일 읽기 */
data c1 ;
infile 'snp_map_hv1' firstobs = 2;
informat name $37. ;
informat hv1chrmo best32. ;
informat hv1pos best32. ;
informat hv1oindex best32. ;
format name $37. ;
format hv1chrmo best32. ;
format hv1pos best32. ;
format hv1oindex best32. ;
input
name
hv1chrmo
hv1pos
hv1oindex
;
hv1index = _n_;
run;
/* 염색체별 SNP 빈도수 */
proc freq data = a1;
table bv2chrmo;
run;
proc freq data = b1;
table bv3chrmo;
run;
proc freq data = c1;
table hv1chrmo;
run;
/* bv2, bv3, hv1 머지하기 */
/* sort */
proc sort data = a1;
by name;
run;
proc sort data = b1;
by name;
run;
proc sort data = c1;
by name;
run;
/* 머지하기 */
data t1;
merge a1 b1 c1;
by name;
run;
/* snp_info 파일 만들기 */
/* 각 snp의 염색체 번호와 위치 저장 */
data t2;
set t1;
/* 각 칩에 없는 SNP index는 0 */
if bv2index = . then bv2index = 0;
if bv3index = . then bv3index = 0;
if hv1index = . then hv1index = 0;
/* 각 SNP의 염색체 번호 */
chr = bv2chrmo;
if bv2chrmo = . then chr = bv3chrmo;
if bv2chrmo = . and bv3chrmo = . then chr = hv1chrmo;
/* 각 SNP의 위치 */
pos = bv2pos;
if bv2pos = . then pos = bv3pos;
if bv2pos = . and bv3pos = . then pos = hv1pos;
/* 각 SNP는 어느 칩에 있는가 */
/* snp_info.txt 만드는 거와 상관은 없지만 그냥 궁금해서 */
if bv2index ~= 0 and bv3index ~= 0 and hv1index ~= 0 then bv2_bv3_hv1 = 'bv2_bv3_hv1';
if bv2index ~= 0 and bv3index ~= 0 and hv1index = 0 then bv2_bv3_hv1 = 'bv2_bv3';
if bv2index ~= 0 and bv3index = 0 and hv1index ~= 0 then bv2_bv3_hv1 = 'bv2_hv1';
if bv2index = 0 and bv3index ~= 0 and hv1index ~= 0 then bv2_bv3_hv1 = 'bv3_hv1';
if bv2index ~= 0 and bv3index = 0 and hv1index = 0 then bv2_bv3_hv1 = 'bv2';
if bv2index = 0 and bv3index ~= 0 and hv1index = 0 then bv2_bv3_hv1 = 'bv3';
if bv2index = 0 and bv3index = 0 and hv1index ~= 0 then bv2_bv3_hv1 = 'hv1';
run;
/* 염색체 번호와 포지션 번호가 잘 들어갔는지 dataset을 열어서 확인 */
proc sort data = t2;
by chr pos;
run;
/* 각 SNP는 어느 비드칩에 있는가. 그냥 궁금해서 */
proc freq data = t2;
table bv2_bv3_hv1 chr * bv2_bv3_hv1 / nocol nopercent;
run;
/* snp_info 출력 */
/* sort */
proc sort data = t2;
by chr pos;
run;
data _null_;
set t2;
file 'snp_info_for_fimpute.txt';
if _n_ = 1 then put "SNP_ID Chr Pos BV3Index HV1Index BV2Index";
put name @41 chr @51 pos @61 bv3index @71 hv1index @81 bv2index ;
run;
/* 제외시킬 SNP 찾기 */
/* 염색체와 위치가 같은 SNP, bv3에 없는 SNP */
/* 30번 이상 염색체는 fimpute에서도 제외 */
/* 염색체 29이하 추출했으나 다시 안 하는 걸로 수정 */
/* 필요없는 부분이나 지우기 뭐 해서 놔 둠 */
data t3;
set t2;
/*
if chr <= 29 then output;
*/
run;
/* 염색체, 위치 순으로 정렬 */
proc sort data = t3;
by chr pos;
run;
/* 염색체 번호와 위치가 같은 SNP 추출 */
data te1;
set t3;
flag1 = 1;
keep chr pos flag1;
if lag(chr) = chr and lag(pos) = pos then output;
run;
data te1_r1;
merge te1 t3;
by chr pos;
if flag1 = 1 then output;
run;
data te1_r2;
set te1_r1;
keep name;
run;
/* bovine version 3에 없는 SNP */
/* bovine version 3로 imputation을 하기 때문.
만일 hanwoo version 1으로 한다면 hv1에 없는 SNP 추출 후 제외 */
data te2;
set t3;
if bv3index = 0 then output;
run;
data te2_r1;
set te2;
keep name;
run;
/* 제외할 SNP 목록 합치기 */
data tef1;
set te1_r2 te2_r1;
run;
/* 중복 SNP 제거 */
proc sort data = tef1;
by name;
run;
data tef2;
set tef1;
by name;
if first.name then output;
run;
/* 출력 */
data _null_;
set tef2;
file "excluded_snp_for_fimpute.txt";
put name;
run;
결과 파일 :
snp_info_for_fimpute.txt


excluded_snp_for_fimpute.txt

illumina2pregs가 만들어 내는 map file의 컬럼 순서가 바뀜에 따라 SAS 프로그램 변경 사항을 추가하였다.
<2021.12.2. 끝.>
<2024.10.28 시작>
일루미나 칩과 Axiom 칩(써모피셔 칩)을 같이 이용해서 imputation을 할 기회가 생겼다. 그런데 문제가 생겼다. 일루미나 칩과 Axiom 칩의 SNP 이름이 서로 다르다는 것이다. 위에서는 SNP의 이름을 기준으로 정렬하고 머지를 했는데 그렇게 할 수가 없다. 그래서 어차피 염색체와 위치 번호를 기준으로 imputation하는 것이므로 한 쪽의 SNP의 이름을 다른 쪽의 SNP 이름으로 바꾸어 imputation 하면 될 것으로 생각했다. 실제로 이렇게 하면 된다. 그런데 약간 복잡했다.
일루미나의 돼지 칩으로 PorcineSNP60v2가 있다. 그리고 국내에서 일루미나 돼지 커스텀 칩을 만들었는데 KPigv2이다. 여기에 써모피셔의 돼지 칩으로 Axiom_PIgHDv1이 있다. 목표는 이 세 칩을 KPigv2로 imputation 하는 것이다.
다음 각 칩의 illumina2pregs를 거친 snp_map이다.
PorcineSNP60v2 (SNP : 61565개)
KPigv2 (SNP : 76756개)
Axiom_PigHDv1 (SNP : 658692개)
그런데 각 칩들을 면밀히 살펴 보면 이상한(?) 점들이 좀 있다. 예를 들어 각 칩별로 SNP 이름은 같은데 염색체와 위치가 다른 SNP들이 있었다. PorcineSNP60v2에서는 36개, KPigv2에서는 212개였다. 그리고 두 개의 일루미나 칩을 합쳐 놓으면 또 문제가 생긴다. 양 칩에서 SNP 이름은 같은데 위치가 다른 SNP가 19개이고, 이름이 다른데 위치가 같은 SNP도 10개 였다. 커스텀 칩을 만들 때 신경을 좀 더 썼어야 하는 것인가? 문제 없는(?) 일루미나 SNP들을 Axiom 칩에 머지할 때도 문제가 생겼다. 염색체와 위치를 기준으로 일루미나 SNP 이름을 붙이는데 Axiom 칩에서 염색체와 위치가 중복되는 SNP들이 있어서 이들이 같은 일루미나 SNP이름으로 대치된다는 점이다. 그러면 snp_info 파일을 만들었을 때 에러가 생긴다. 방법은 머지할 때 염색체와 위치가 중복될 때 그 중 하나만 일루미나 SNP 이름으로 대치되게 해야 한다. 암튼 일루미나 칩을 다루는 것보다, Axiom 칩 하나 추가된다고 해야 할이 일이 굉장히 많아 진다는 점이다. 다음은 snp_info 파일을 만들고 imputation에서 제외될 snp 목록을 만드는 SAS 프로그램이다.
/*
file name : make_porcine_snp_info_for_fimpute.sas
FImpute 프로그램을 위한 porcine_snp_info.txt 파일 만들기
PorcineSNP60 v2 BeadChip
Axiom Porcine Genotyping Array (Axiom_PigHDv1) -> KPigv2로 imputation할 때 필요한 porcine_snp_info.txt 만들기
*/
/****************************/
/* KPigv2 snp_map 파일 읽기 */
/****************************/
data a1 ;
infile '../01_data/snp_map_kpigv2' firstobs = 2;
informat name $37. ;
informat kpigv2chrmo best32. ;
informat kpigv2pos best32. ;
informat kpigv2oindex best32. ;
input
name
kpigv2chrmo
kpigv2pos
kpigv2oindex
;
kpigv2index = _n_;
run;
/* no of data : 76756 */
/* 염색체별 snp 빈도 */
proc freq data = a1;
table kpigv2chrmo;
run;
/* 1 ~ 18 염색체 중 chrmo와 pos가 중복되는 snp */
proc sort data = a1;
by kpigv2chrmo kpigv2pos;
run;
data a1_e1;
set a1;
if kpigv2chrmo > 18 then delete;
if lag(kpigv2chrmo) = kpigv2chrmo and lag(kpigv2pos) = kpigv2pos then output;
keep kpigv2chrmo kpigv2pos;
run;
/*
no of data : 131
*/
/* 여기서도 중복이 있어 중복 제거 */
data a1_e2;
set a1_e1;
by kpigv2chrmo kpigv2pos;
if first.kpigv2pos;
run;
/*
no of data : 81
*/
/* 문제있는 snp 추출 */
data a1_e3;
merge a1 (in = x1) a1_e2 (in = x2);
by kpigv2chrmo kpigv2pos;
if x2;
run;
/*
no of data : 212
*/
PROC EXPORT DATA= A1_e3
OUTFILE= "kpigv2_snp_in_trouble.xlsx"
DBMS=EXCEL REPLACE;
SHEET="kpigv2_snp_in_trouble";
RUN;
/* name 변수만 남기기 */
data a1_e3_name;
set a1_e3;
keep name;
run;
/* 1 - 18까지 염색체 중 문제없는 snp 추출 */
data a2;
merge a1 (in = x1) a1_e2 (in = x2);
by kpigv2chrmo kpigv2pos;
if x2 or kpigv2chrmo > 18 then delete;
run;
/*
no of data : 71806
*/
/************************************/
/* PorcineSNP60v2 snp_map 파일 읽기 */
/************************************/
data b1 ;
infile '../01_data/snp_map_porcv2' firstobs = 2;
informat name $37. ;
informat porcv2chrmo best32. ;
informat porcv2pos best32. ;
informat porcv2oindex best32. ;
input
name
porcv2chrmo
porcv2pos
porcv2oindex
;
porcv2index = _n_;
run;
/* no of data : 61565 */
/* 염색체별 snp 빈도 */
proc freq data = b1;
table porcv2chrmo;
run;
/* 1 ~ 18 염색체 중 chrmo와 pos가 중복되는 snp */
proc sort data = b1;
by porcv2chrmo porcv2pos;
run;
data b1_e1;
set b1;
if porcv2chrmo > 18 then delete;
if lag(porcv2chrmo) = porcv2chrmo and lag(porcv2pos) = porcv2pos then output;
keep porcv2chrmo porcv2pos;
run;
/*
no of data : 18
*/
/* 여기서도 중복이 있다면 중복 제거 */
data b1_e2;
set b1_e1;
by porcv2chrmo porcv2pos;
if first.porcv2pos;
run;
/*
no of data : 18
*/
/* 문제있는 snp 추출 */
data b1_e3;
merge b1 (in = x1) b1_e2 (in = x2);
by porcv2chrmo porcv2pos;
if x2;
run;
/*
no of data : 36
*/
PROC EXPORT DATA= b1_e3
OUTFILE= "porcv2_snp_in_trouble.xlsx"
DBMS=EXCEL REPLACE;
SHEET="porcv2_snp_in_trouble";
RUN;
/* name 변수만 남기기 */
data b1_e3_name;
set b1_e3;
keep name;
run;
/* 1 - 18까지 염색체 중 문제없는 snp 추출 */
data b2;
merge b1 (in = x1) b1_e2 (in = x2);
by porcv2chrmo porcv2pos;
if x2 or porcv2chrmo > 18 then delete;
run;
/*
no of data : 58828
*/
/******************************************/
/* kpigv2와 porcv2의 문제 없는 snp들 머지 */
/******************************************/
proc sort data = a2;
by name;
run;
proc sort data = b2;
by name;
run;
data s1;
merge a2 b2;
by name;
run;
/*
no of data : 74596
*/
/* 이름은 같은데 염색체, 위치가 다른 snp 추출 */
data s1_e1;
set s1;
if (kpigv2chrmo ~= . and porcv2chrmo ~= . )
and (kpigv2chrmo ~= porcv2chrmo or kpigv2pos ~= porcv2pos) then output;
run;
/*
no of data : 19
*/
PROC EXPORT DATA= s1_e1
OUTFILE= "illumina_porcine_snp_same_name_dif_pos.xlsx"
DBMS=EXCEL REPLACE;
SHEET="illumina_porcine_snp_in_trouble";
RUN;
/* name 변수만 남기기 */
data s1_e1_name;
set s1_e1;
keep name;
run;
/* 이름은 같은데 염색체, 위치가 다른 snp를 제거한 snp list */
data s2;
set s1;
chrmo = kpigv2chrmo;
if kpigv2chrmo = . then chrmo = porcv2chrmo;
pos = kpigv2pos;
if kpigv2pos = . then pos = porcv2pos;
if (kpigv2chrmo ~= . and porcv2chrmo ~= . )
and (kpigv2chrmo ~= porcv2chrmo or kpigv2pos ~= porcv2pos) then delete;
keep name chrmo pos;
run;
/*
no of data : 74577
*/
/* 기우에 따라 한 번 더 점검 : unique name */
proc sort data = s2;
by name;
run;
data s2_e1;
set s2;
by name;
if not first.name;
run;
/*
no of data : 0
*/
/* 기우에 따라 한 번 더 점검 : unique chrmo and pos */
proc sort data = s2;
by chrmo pos;
run;
data s2_e2;
set s2;
by chrmo pos;
if not first.pos;
keep chrmo pos;
run;
/*
no of data : 5, 기우가 아니네!
*/
/* 문제 있는 snp 추출 */
data s2_e3;
merge s2 (in = x1) s2_e2(in = x2);
by chrmo pos;
if x2;
run;
/*
no of data : 10
*/
PROC EXPORT DATA= s2_e3
OUTFILE= "illumina_porcine_snp_dif_name_same_pos.xlsx"
DBMS=EXCEL REPLACE;
SHEET="illumina_porcine_snp_in_trouble";
RUN;
/* name 변수만 남기기 */
data s2_e3_name;
set s2_e3;
keep name;
run;
/* 문제 없는 snp 추출 */
data s3;
merge s2 (in = x1) s2_e2(in = x2);
by chrmo pos;
if x2 then delete;
run;
/*
no of data : 74567
*/
/********************************************************************/
/* Axiom Porcine Genotyping Array (Axiom_PigHDv1) snp_map 파일 읽기 */
/********************************************************************/
data c1 ;
infile '../01_data/snp_map_apigv1' firstobs = 2;
informat name1 $37. ;
informat apigv1chrmo best32. ;
informat apigv1pos best32. ;
informat apigv1oindex best32. ;
input
name1
apigv1chrmo
apigv1pos
apigv1oindex
;
chrmo = apigv1chrmo;
pos = apigv1pos;
apigv1index = _n_;
run;
/* no of data : 658692 */
/***********************************************/
/* 이제 axiom pig v1에 illumina snp_id 를 머지 */
/***********************************************/
/* sort */
proc sort data = c1;
by chrmo pos;
run;
proc sort data = s3;
by chrmo pos;
run;
/* merge */
data c2;
merge c1 (in = x1) s3 (in = x2);
by chrmo pos;
/* apigv1에서 염색체, 위치가 중복되면
머지되는 이름도 중복되는데
그러면 에러라서 처음 하나만 머지하고 나머지는 empty */
if not first.pos then name = '';
if x1 then output;
run;
/* 몇 개의 snp_id가 붙었는지 확인 */
data c2_2;
set c2;
if name ~= '' then output;
run;
/*
no of data : 54265,
illumina snp_id 74567개 중 54265개가 붙었다.
*/
/* illumina snp_id를 가진 axiom snp_id 출력 */
PROC EXPORT DATA= c2_2
OUTFILE= "apigv1_illumina_porcine.xlsx"
DBMS=EXCEL REPLACE;
SHEET="apigv1_illumina_porcine";
RUN;
data c3;
set c2;
if name = '' then name = name1;
keep name apigv1chrmo apigv1pos apigv1oindex apigv1index;
run;
/*
axiom pig v1의 snp_id를 illumina snp_id로 대체 완료, 658962
*/
/* 염색체 18 이하 중 chromosome, postion이 중복되는 snp */
proc sort data = c3;
by apigv1chrmo apigv1pos;
run;
data c3_e1;
set c3;
if apigv1chrmo > 18 then delete;
if lag(apigv1chrmo) = apigv1chrmo and lag(apigv1pos) = apigv1pos then output;
keep apigv1chrmo apigv1pos;
run;
/*
no of data : 28
*/
/* 염색체 18 이하 중 chromosome, postion이 중복되는 snp 쌍 */
data c3_e2;
merge c3 (in = x1) c3_e1 (in = x2);
by apigv1chrmo apigv1pos;
if x2;
run;
/*
no of data : 56
*/
PROC EXPORT DATA= c3_e2
OUTFILE= "apigv1_snp_in_trouble.xlsx"
DBMS=EXCEL REPLACE;
SHEET="apigv1_snp_in_trouble";
RUN;
/* name 변수만 남기기 */
data c3_e2_name;
set c3_e2;
keep name;
run;
/*
no of data : 56
*/
/**********************************************************/
/* 이제 kpigv2 porcv2 apigv1을 이름으로 정렬하고 머지하기 */
/**********************************************************/
/* sort */
proc sort data = a1;
by name;
run;
proc sort data = b1;
by name;
run;
proc sort data = c3;
by name;
run;
/* merge */
data t1;
merge a1 b1 c3;
by name;
run;
/* no of data : 684064 */
data t2;
set t1;
/* 없었던 SNP index는 0 */
if kpigv2index = . then kpigv2index = 0;
if porcv2index = . then porcv2index = 0;
if apigv1index = . then apigv1index = 0;
/* 각 SNP의 염색체 번호 */
chr = kpigv2chrmo;
if kpigv2chrmo = . then chr = porcv2chrmo;
if kpigv2chrmo = . and porcv2chrmo = . then chr = apigv1chrmo;
/* 염색체 번호 100 -> 24 */
if chr = 100 then chr = 24;
/* 각 SNP의 위치 */
pos = kpigv2pos;
if kpigv2pos = . then pos = porcv2pos;
if kpigv2pos = . and porcv2pos = . then pos = apigv1pos;
run;
/* no of data : 684064 */
/* chromosome 별 snp 개수 그냥 확인 */
proc freq data = t2;
table chr;
run;
/* name이 unique한지 검사 */
proc sort data = t2;
by name;
run;
data t2_e1;
set t2;
by name;
if not first.name;
run;
/*
no of data : 0
*/
/* snp_info 출력 */
/* sort */
proc sort data = t2;
by chr pos;
run;
data _null_;
set t2;
file 'snp_info_for_porcine_fimpute.txt';
if _n_ = 1 then put "SNP_ID Chr Pos kpigv2Index porcv2Index apigv1Index";
put name @41 chr @51 pos @61 kpigv2index @71 porcv2index @81 apigv1index ;
run;
/* no of data : 684064 */
/***************************************/
/* 제외시킬 SNP 찾기 */
/* 18번 초과 염색체는 fimpute에서 제외 */
/***************************************/
/* 기준 칩 kpigv2에 없는 염색체 18번 이하 snp 추출 */
data x1_name;
set t2;
if chr <= 18 and kpigv2index = 0 then output;
keep name;
run;
/*
no of data : 540542
*/
/* 문제가 있는 snp 이름 모으기 */
data x2;
set
a1_e3_name /* kpigv2 염색체 위치 중복 snp */
b1_e3_name /* porcv2 염색체 위치 중복 snp */
s1_e1_name /* kpigv2 porcv2, same name, different pos */
s2_e3_name /* kpigv2 porcv2, different name, same pos */
c3_e2_name /* apigv1 염색체 위치 중복 snp */
x1_name; /* snp of apigv1 not in kpigv2 */
run;
/*
no of data : 540875
*/
/* 중복 SNP 제거 */
proc sort data = x2;
by name;
run;
data x3;
set x2;
by name;
if first.name then output;
run;
/* no of data : 540783 */
/* 출력 */
data _null_;
set x3;
file "excluded_snp_for_porcine_fimpute.txt";
put name;
run;
암튼 이렇게 snp_info 파일을 만들어 imputation을 하니 된다. 그런데 어차피 일루미나 KPigv2로 imputation하는 거라면 Axiom_PigHDv1의 70만개 SNP를 출력하지 말고, KPigv2와 공통된 SNP들만 다운로드하는 것도 방법일 것이다. 그리고 다운로드할 때 일루미나 SNP 이름으로 다운로드 한다면 마치 일루미나 칩들을 imputation하는 것처럼 비교적 간단히 imputation 할 수 있을 것으로 보인다.
마지막으로 경험에서 우러나온 얘기 한 마디. snp_info 파일을 만들었으면 반드시 snp_info 파일이 잘 만들어졌는지 확인해야 한다. 확인하는 방법은 snp_id 가 unique한지, 각 칩의 index가 0을 제외하고 unique한지 보는 것이다. FImpute가 snp_info 파일이 이상하다고 프로그램이 중단되는데 그 에러 메시지가 그리 친절하지 않다. 에러 메시지를 보고 원인을 찾기 힘들다. 그럴 때 snp_id, 각 칩의 index가 unique한지 점검하기 바란다. 프로그램을 실행하기 전에 점검하는 것이 시간을 절약하는 길이다. (그러나 이렇게 얘기를 해도 점검하는 사람을 못 봤다. 결국 이해할 수 없는 프로그램 에러를 보고 몇날 며칠을 고민하다 점검한다. 나도 그렇다. 인간의 어리석음이란 ...)
<2024.10.28. 끝.>
■ 혈통 파일 준비
다음과 같이 혈통 파일을 준비한다.

Male이 dam 열에 나타나거나, Female이 sire 열에 나타나면 에러를 일으키므로 사전에 검토하는 것이 바람직하다.
■ control 파일의 준비
control 파일은 준비한 파일의 위치와 이름, imputation 옵션, 출력 폴더 등을 지정하는 파일이다. 다음과 같은 control 파일을 준비한다.
| title="BovineSNP50v3 -> HanwooSNP50v1 imputation"; genotype_file="HanwooSNP50v1_for_fimpute_01.txt" "BovineSNP50v3_for_fimpute_01.txt"; snp_info_file="snp_info_for_fimpute.txt"; ped_file="pedigree.txt"; output_folder="output1"; exclude_snp = "excluded_snp_for_fimpute.txt"; exclude_chr = 30 31 32 33; //parentage_test /ert_mm=0.02 /remove_conflict; save_genotype; njob=6; |
control 파일 설명은 다음과 같다.
title="BovineSNP50v3 -> HanwooSNP50v1 imputation";
- imputation 제목
genotype_file="HanwooSNP50v1_for_fimpute_01.txt" "BovineSNP50v3_for_fimpute_01.txt";
- imputation에 사용할 genotype file
snp_info_file="snp_info_for_fimpute.txt";
- snp_info 파일
ped_file="pedigree.txt";
- 혈통 파일
output_folder="output1";
- 결과를 출력할 폴더
exclude_snp = "excluded_snp_for_fimpute.txt";
- imputation에서 제외할 snp 이름이 들어있는 파일
exclude_chr = 30 31 32 33;
- imputation에서 제외할 염색체 번호
//parentage_test /ert_mm=0.02 /remove_conflict;
- 친자감정을 할 수 있음. 여기서는 무시
save_genotype;
- haplotype이 아니라 genotype을 저장
njob=6;
- 6개의 process를 이용하여 작업 수행. cpu의 core가 많을 경우 그에 따라 증가
■ FImpute의 실행
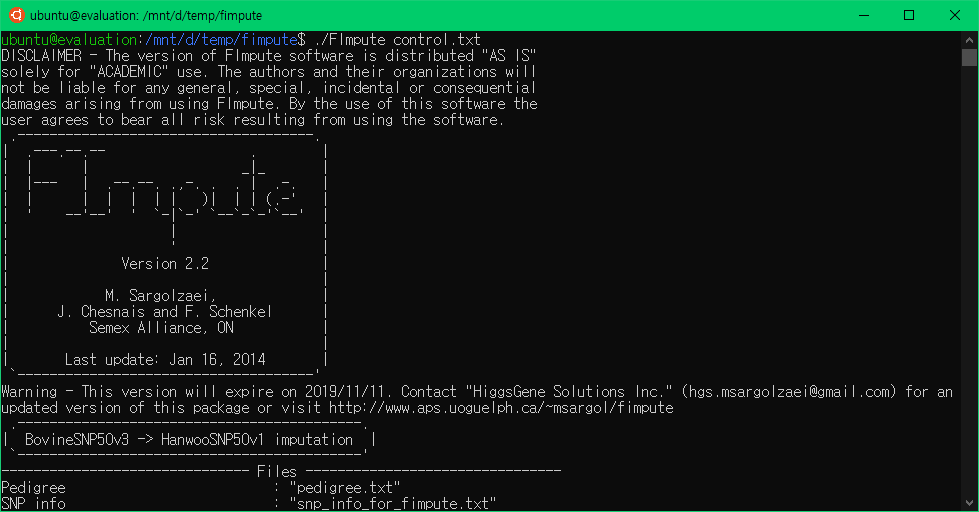
리눅스에서만 FImpute를 실행할 수 있다. 실행하기 위하여 필요한 파일을 리눅스 컴퓨터의 폴더에 복사한다. 여기서는 윈도우즈 10의 Windows Subsystem for Linux 중 Ubuntu를 설치하고 실행한 화면이다.

imputation에 필요한 파일을 볼 수 있다. 다음은 FImpute를 실행한 화면이다.






output1 폴더에 imputation된 결과를 볼 수 있다. genotypes_imp.txt 파일이다.

새로운 snp_info파일이 생성되었는데 다음과 같다. 51857개의 snp가 최종적임을 알 수 있다.


■ genotype 파일 되돌리기
imputation 된 genotype 파일에는 header와 chip 번호가 있어서 이것을 제거해야 한다. header와 chip 번호를 제거하는 awk 프로그램이다.
delete_chip_number_50k.awk
| BEGIN{ } { if( NR >= 2 )print $1, $3 > "50k_for_gs.txt" } END { } |
2행 이상에 대해 1열과 3열을 출력.
실행을 간편하게 하기 위하여 다음과 같이 배치 파일을 작성한다.
run_delete_chip_number_50k.awk.bat
| awk -f delete_chip_number_50k.awk ./output1/genotypes_imp.txt pause |
awk 프로그램 이름과 처리하려는 파일 이름
윈도우즈 탐색기에서 run_delete_chip_number_50k.awk.bat 파일을 더블클릭하여 실행할 수 있다. 다음은 실행 화면이다.

결과적으로 생성된 파일은 다음과 같다.

■ snp_map 파일 되돌리기
output1 폴더의 snp_info.txt의 1행을 삭제하고 열 순서를 바꾸어 준다. 다음과 같은 awk 프로그램을 이용한다.
make_snp_map_after_fimpute.awk
| BEGIN{ } { if( NR >= 2 ) print $4, $2, $3, $1, $4> "snp_map_for_gs.txt" } END { } |
2행 이상에 대하여 4열, 2열, 3열, 1열, 다시 4열 순으로 snp_map_for_gs.txt 파일에 쓰기
위 프로그램을 실행하기 편하게 하기 위하여 다음과 같은 배치 파일을 만든다.
run_make_snp_map_after_fimpute.awk.bat
| awk -f make_snp_map_after_fimpute.awk ./output1/snp_info.txt pause |
윈도우즈 탐색기에서 run_make_snp_map_after_fimpute.awk.bat 파일을 더블클릭하여 실행할 수 있다. 다음은 실행 화면이다.

프로그램 실행 후 결과 파일은 다음과 같다.

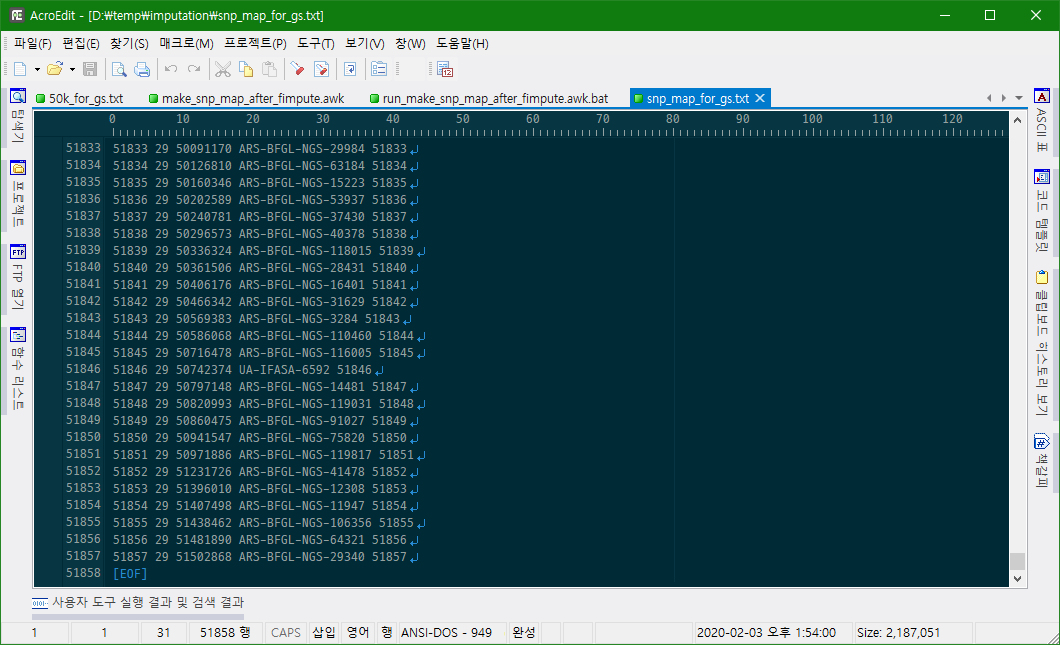
<2021.12.2. 시작>
illumina2pregs가 만들어 내는 map file 형식이 바뀌었다. fimpute 가 만들어낸 snp_info.txt 파일을 가지고 illumina2pregs가 만들어 낸 것과 같은 형식의 map file을 만들어야 한다.
다음은 bovine version 2, bovine version 3, hanwoo version 1을 bovine version 3로 imputation한 결과의 snp_info.txt이다.

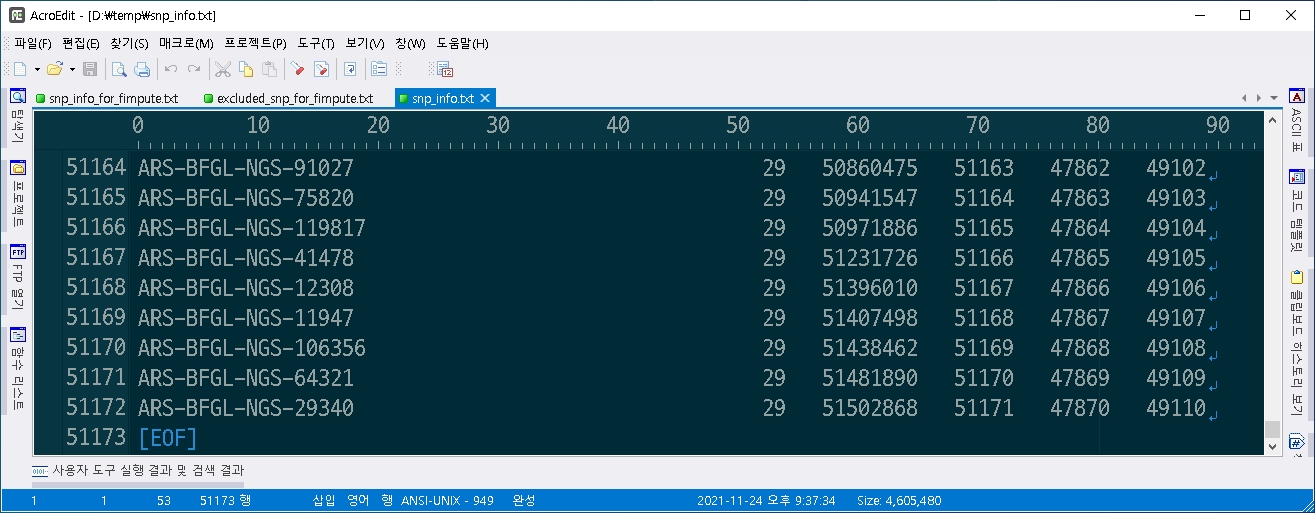
염색체 29번까지 남았다. 총 51171개의 SNP만 남았다.
전과 차이가 나는 것은 header가 있고, 순서가 snp_id(이름), chr(염색체), pos(위치), index(순서)이다. 위 snp_info파일을 그대로 써도 문제 없을 것 같고, chip_1의 index만 남기고 싶으면 다음과 같이 한다.
make_snp_map_after_fimpute.awk
BEGIN{
}
{
print $1, $2, $3, $4 > "snp_map_for_gs.txt"
}
END {
}run_make_snp_map_after_fimpute.awk.bat 파일을 만들어 실행을 쉽게 한다.
awk -f make_snp_map_after_fimpute.awk ./output1/snp_info.txt
pauseoutput1 폴더의 snp_info.txt 파일을 재료로 하여 make_snp_map_after_fimpute.awk를 실행한다.윈도우 탐색기에서 run_make_snp_map_after_fimpute.awk.bat 을 더블 클릭하여 실행할 수 있다.

실행 결과 생긴 snp_map_for_gs.txt


<2021.12.2. 끝>
서로 다른 버전의 coded SNP genotype과 snp_map을 이용하여 하나의 SNP genotype과 snp_map을 만들었다. 다음은 이것을 유전능력 평가에 이용하는 것이다.
Advice : Call rate 가 낮은 개체의 유전자형을 포함하여 분석을 하지 마세요. 처음에는 call rate가 낮아도 imputation하면 되겠지라고 생각했는데 역시 되지 않습니다.. call rate가 낮은면 call 된 유전자형도 믿을 수가 없고 믿을 수 없는 유전자형을 이용하면 전체 imputation이 엉망이 될 것입니다. 경험적으로 90% 미만이든, 이하든 사용하지 않는게 정신건강에 좋을 것입니다.
FImpute : 이게 유료화 되었습니다. 새로운 버전이 업로드 안되어 메일을 쓰니 유료화되었답니다. 일년에 몇 번 안하는 거면 컴퓨터 날짜를 바꾸시고, 주기적으로 업무에 사용하시는 거라면 돈주고 사시는 것이 좋을 것 같습니다. 저도 안 사 봐서 얼마인지 모르겠습니다. 윈도우즈에서 wsl2를 설치하시고, 윈도우즈 설정의 날짜 및 시간 자동으로 하는 거 좀 꺼 주시고, wsl2에서 date 명령으로 날짜를 바꾸시면 시간이 지난 FImpute가 ... 정상적인 방법이 아니니 하라 말라 못하겠습니다.
'Animal Breeding > Genomic Selection' 카테고리의 다른 글
| QCF90이용한 quality control (0) | 2020.02.05 |
|---|---|
| PREGSF90을 이용한 Quality Control (0) | 2020.02.04 |
| illumina2pregs로 SNP genotype을 0, 1, 2로 변환하기 (0) | 2020.01.09 |
| GenomeStudio FinalReport 만들기 (1) | 2020.01.08 |
| 지놈스튜디오 파일(BSC 파일) 만들기 (1) | 2020.01.06 |