Illumina BovineSNP50 v3로 SNP Genotyping을 하면 약 5만 개 SNP의 genotype 결과를 얻을 수 있다.

자세한 실험과정은 생략하더라도 실험의 마지막 과정은 BeadChip을 스캔하고, 스캔한 결과 나온 이미지 파일을 GenomeStudio로 읽어서 최종 Genotype을 얻게 되는데 GenomeStudio로 스캔한 이미지를 읽은 결과가 확장자가 bsc인 지놈스튜디오 파일이다. 물론 genotyping을 한 실험실에서 최종적인 genotype을 알려주긴 하나, 스캔 이미지를 읽어 확장자가 bsc인 지놈스튜디오 파일을 만드는 과정을 살펴보고 최종적인 genotype을 어떻게 출력하는지 살펴봄으로써 genotype을 관리하는 팁을 구할 수 있을 것이다.
실험과정 마지막에 BeadChip을 iScan으로 스캔하면 이미지 파일이 생성된다.

스캔하여 결과로 나온 이미지 파일은 다음과 같다.
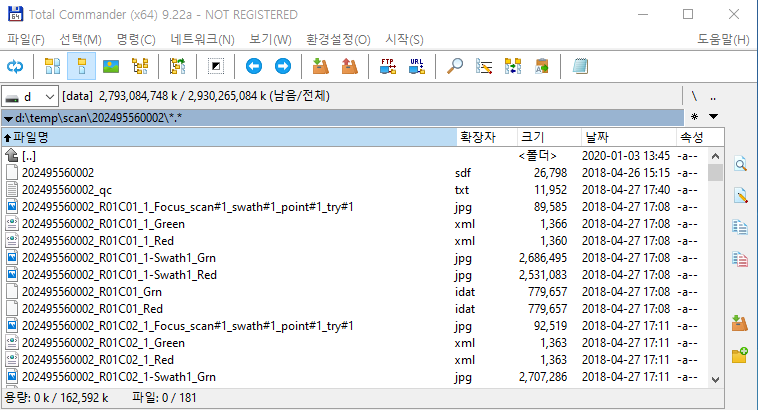
폴더 이름이 202495560002 인데 이것은 칩의 고유번호로 보인다. 파일명을 보면 칩 고유번호에 행 번호와 열 번호로 된 것임을 알 수 있다. 현재 칩은 한 번에 24두를 분석하는 것으로 행이 12까지 열이 2까지 있다. 칩의 하나의 셀에 대하여 총 7개의 파일이 생긴 것을 볼 수 있다. 24두를 분석하면 최소 24 x 7 = 168개의 파일과 그 외 몇 가지 파일이 더 생긴다. 이제 이들 이미지를 이용해서 genotype을 만들어 내야 하는데 먼저 GenomeStudio가 이들 이미지 파일을 읽어야 한다.
GenomeStudio로 이미지 파일을 읽어 들일 때는 세 가지 파일이 더 필요하다.
sample sheet : 위 이미지 파일을 보면 행열 번호만 나와 있을 뿐 이게 누구의 genotype인지 안 나와 있다. sample sheet에는 몇 행 몇 열은 누구의 것이다라는 정보가 포함되어 있다. 없어도 이미지를 읽을 수는 있으나 누구의 것인지 알 수 없다.
Beed Pool Manifest files(.bpm) : 확장자가 bpm인 이 파일은 이 칩이 무슨 칩인지 알려주는 파일이다. 즉 소 BeadChip인지, 돼지 BeadChip인지 알려주고, 소 BeadChip에서 몇 번째 버전인지 알려주는 파일이다. 없으면 안 된다. 일루미나 홈페이지에서 다운로드 받거나 Genotyping한 실험실에서 제공받을 수 있다.
Cluster files(.egt) : 한 치의 오류도 없이 정확히 genotype을 알아낼 것 같지만 사실 그렇지 않다. 스캔한 이미지를 보고서 A, T, G, C를 구분하는데 경계가 명확하기도 하고, 안 그렇기도 하다. 또는 Auto Clustering을 할 경우 하나의 칩을 읽을 때와 여러 개의 칩을 한꺼번에 GenomeStudio로 읽으면 결과가 달라질 수 있다. 그걸 방지하고자 기준을 정한 것이 Cluster file이다. 이 파일 없으면 이미지를 읽고 auto clustering 하면 된다.
여기서는 먼저 sample sheet와 cluster file이 없고, bpm 파일만 있다고 가정하고 시작한다. 일루미나 홈페이지에서 GenomeStudio를 다운로드 받아 설치하고 실행한다.
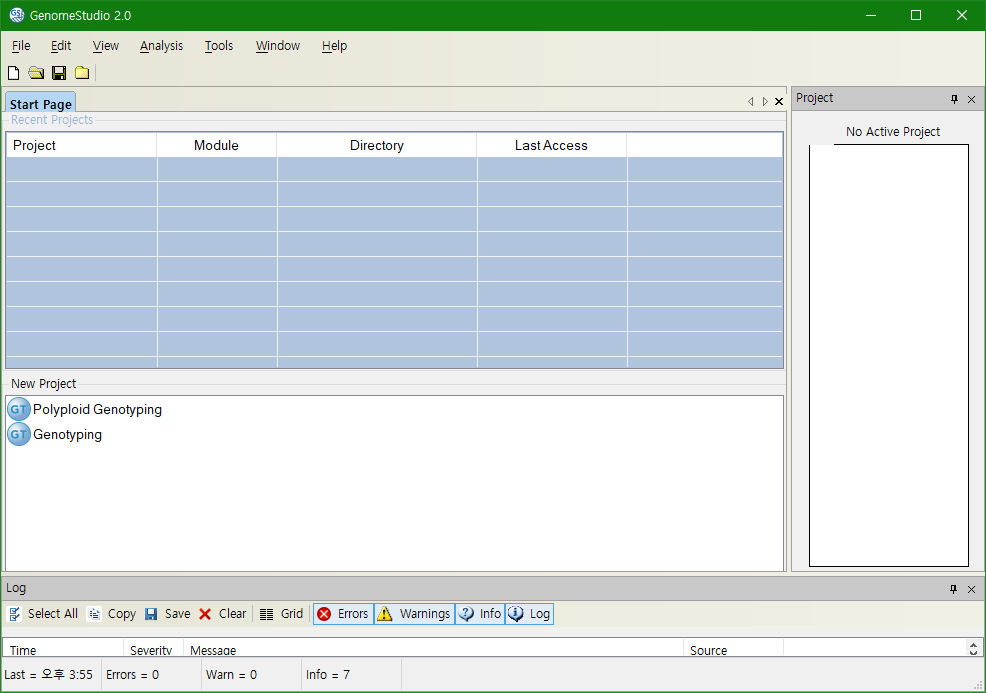
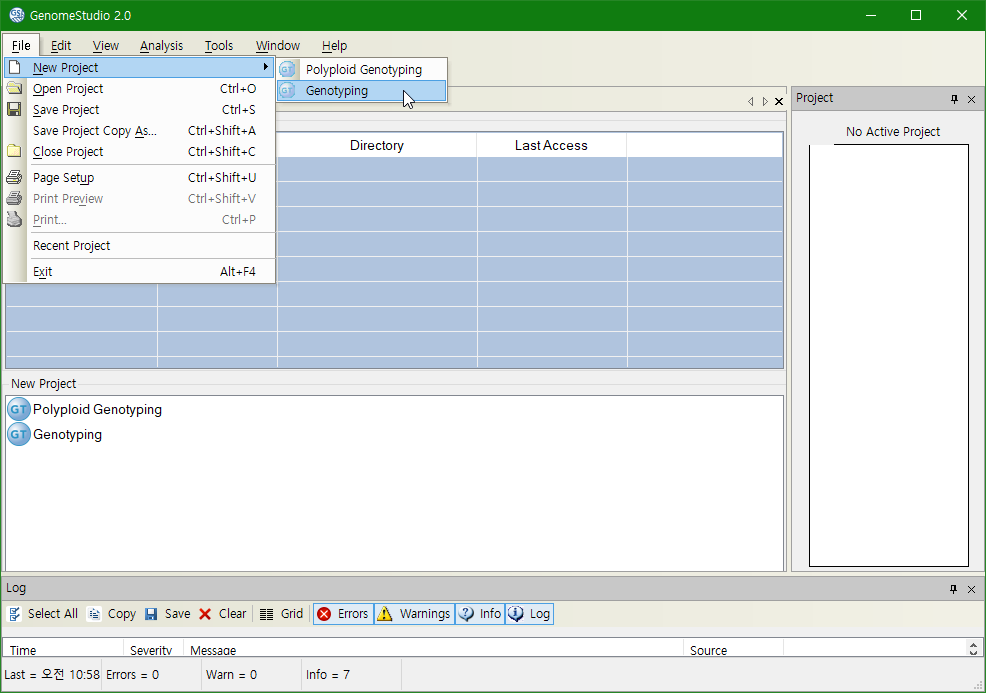
File – New Project – Genotyping 클릭
Next 클릭

Projects Repository에 지놈스튜디오 파일을 저장한 폴더를 지정하고, Project Name을 입력한다. Next를 클릭한다.
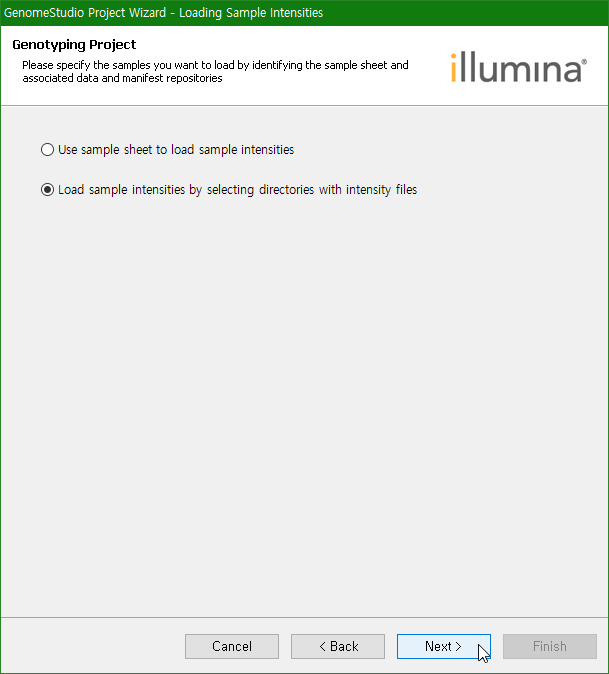
sample sheet를 가지고 있지 않으므로 ‘Load sample intensities ...’를 선택하고 Next를 클릭한다.
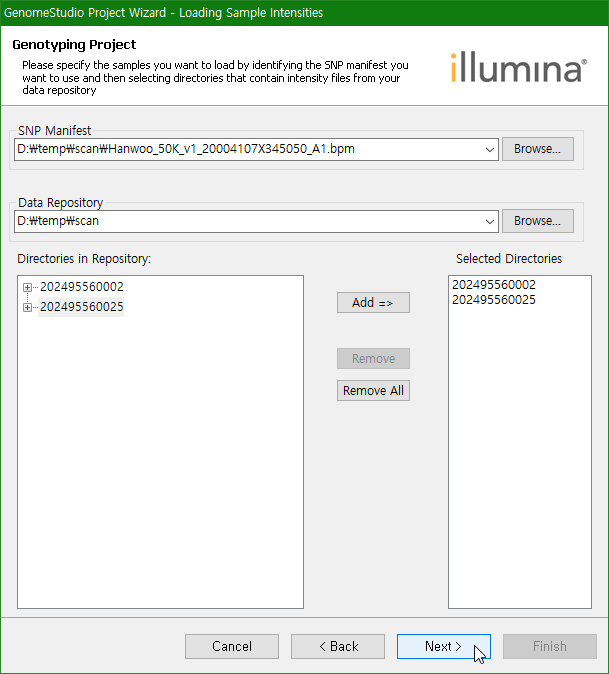
bpm 파일을 지정하고, 스캔한 칩이 있는 폴더를 지정한다. 여기서는 두 개의 칩을 읽어 총 48두의 이미지를 읽을 것이다. GenomeStudio에서 읽을 칩을 선택하고 Add를 클릭한다. Next를 클릭하다.
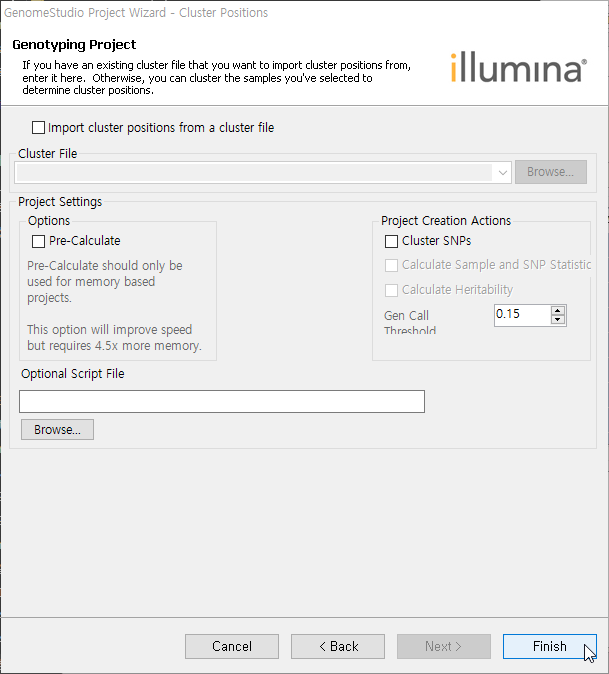
Cluster file을 선택할 수 있는 화면인데 없다고 가정했으므로 그냥 Finish 클릭한다.
이미지를 읽고 있는 중이다.
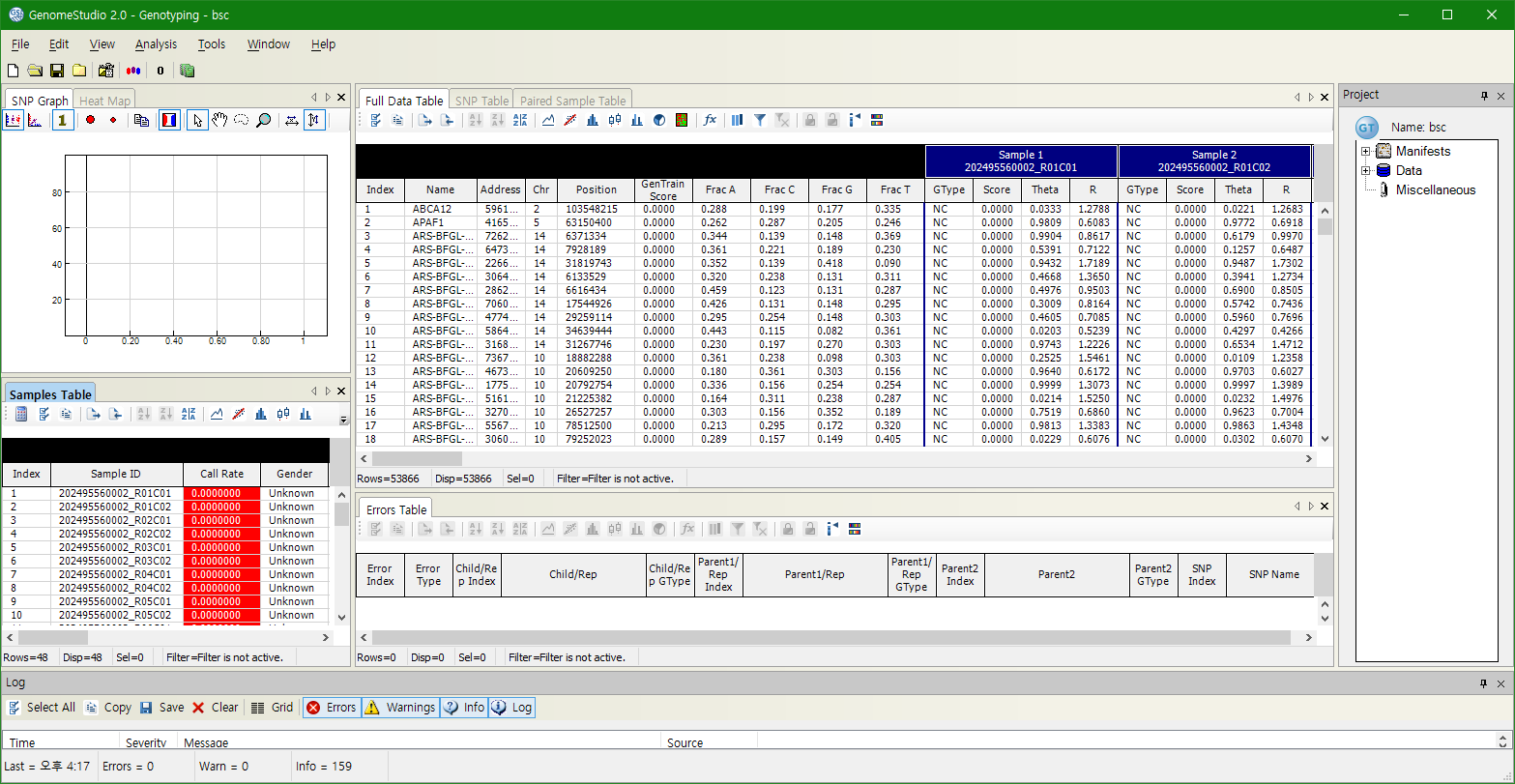
왼쪽 아래 창에서 48두를 읽은 것을 확인할 수 있으나 누구의 것인지 알 수가 없다. 중앙의 창에는 SNP 목록이 나오지만 각 샘플들의 Genotype이 결정되지 않았음을 알 수 있다.
먼저 Genotype을 결정하자. Analysis – Cluster All SNPs 클릭한다.


클러스터 진행 중

모든 SNP에 대한 통계량 업데이트
진행 중
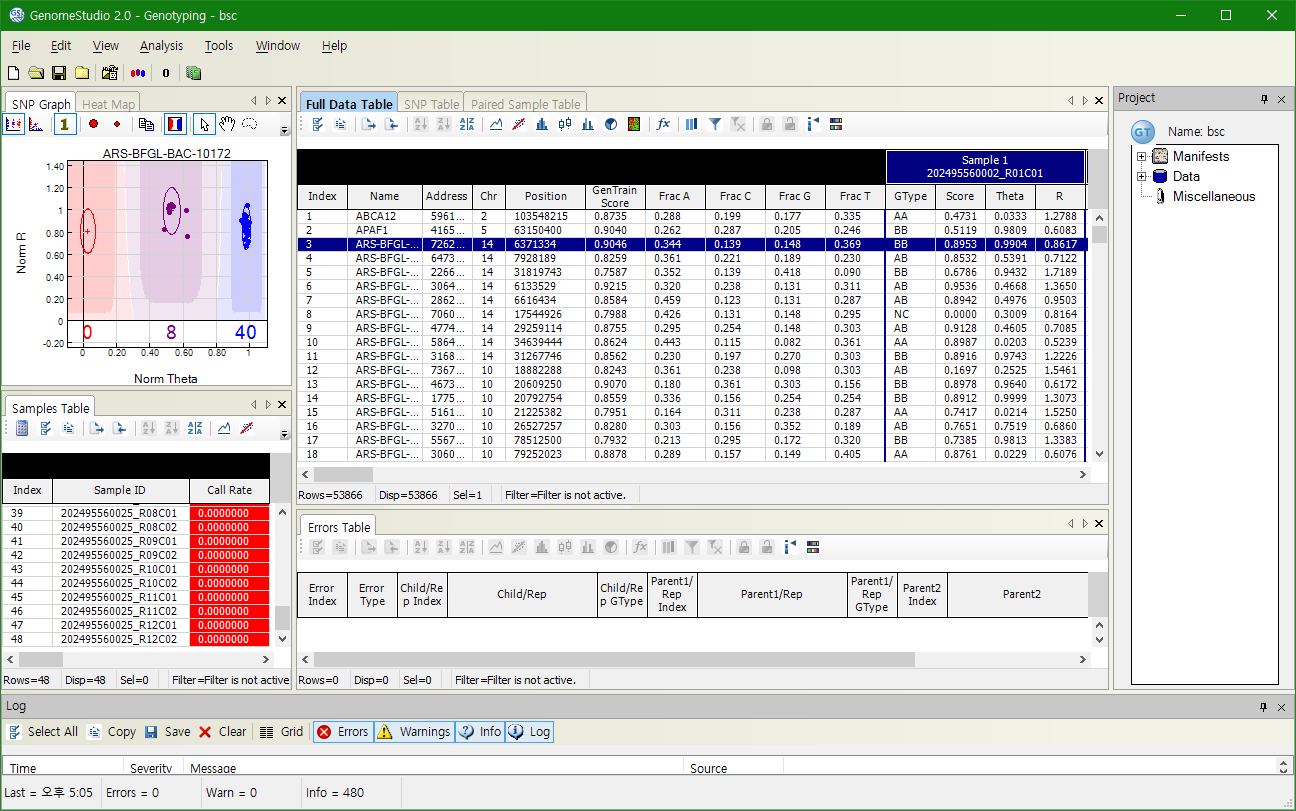
Sample 1의 Genotype이 결정되었음을 알 수 있고, 예를 들어 3번 SNP를 클릭했을 때 왼쪽 위의 창에 클러스터링이 어떻게 되어 있는지도 나와 있다.
이 기준을 다음에도 적용하고 싶다면 egt 파일을 만들고 다른 칩에 적용하면 된다.
File – Export Cluster Positions – For All SNPs를 클릭한다.
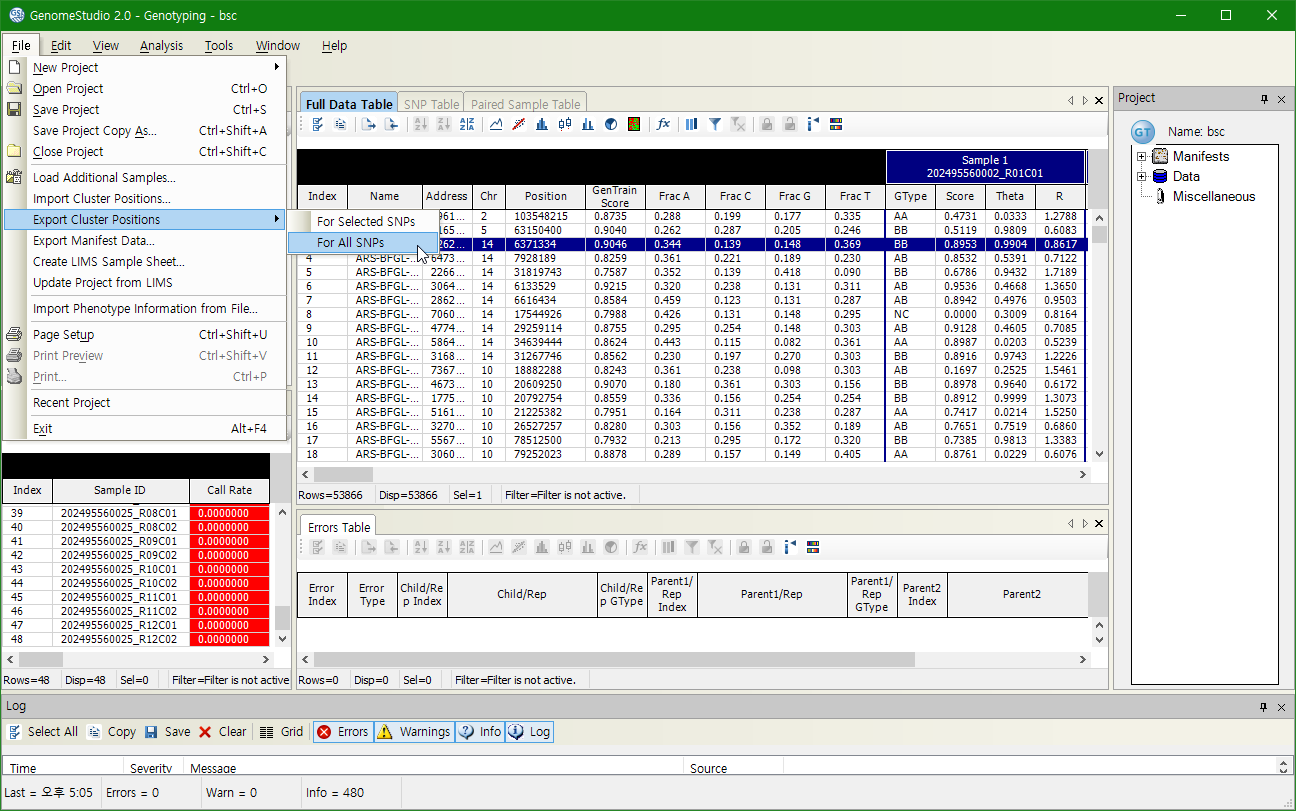

생성된 egt 파일을 확인할 수 있다.
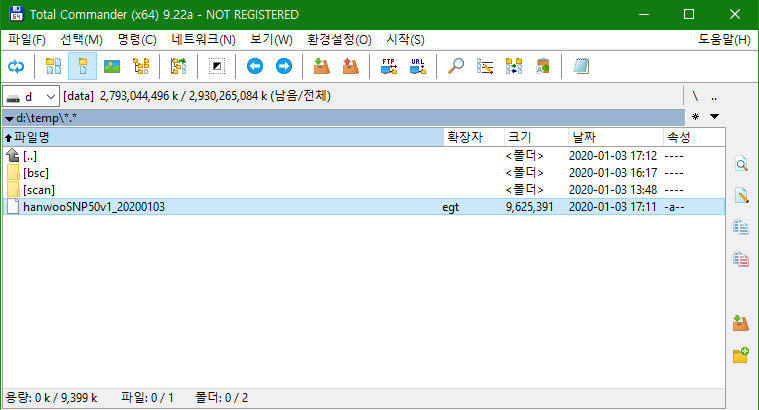
clustering 기준은 품종마다, BeadChip 버전마다 다를 수 있으므로 새로운 품종, 새로운 버전이 있을 경우 많은 개체를 genotyping하여 기준이 되는 egt 파일을 생성하고 그것을 계속 사용하는 것이 좋을 것으로 보인다. 일루미나에서는 집단을 대표하는, 암 수 골고루, nontumor이고 염색체 이상이 없는, call rate 상당히 높은(상당히 높으려면 털에서 DNA 추출하지 말고 혈액 또는 조직에서 추출하자. 상당히 높다는 것이 주관적인데 0.99면 되지 않을까), 수 백 마리(하한은 있는데 상한이 있는지 모르겠다. 600에서 800이면 될 듯)의 자료를 이용하여 egt 파일을 만들 것을 권고하고 있다. 이거 되게 중요한 건데 신경쓰는 사람을 본 적이 없다. 몰라서 그렇다. 알면 실천하자.
그러나 아직도 이 genotype이 누구의 genotype인지 알 수가 없다. 누구의 genotype인지는 sample sheet에 넣는다고 했는데 그것을 알아보자.
지놈스튜디오 프로젝트 파일을 저장한 폴더로 이동해 보자.
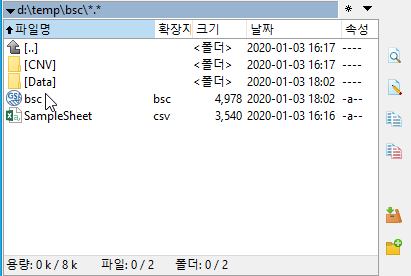
SampleSheet.csv 라는 파일이 생성된 것을 볼 수 있다. 파일을 열어 보자.
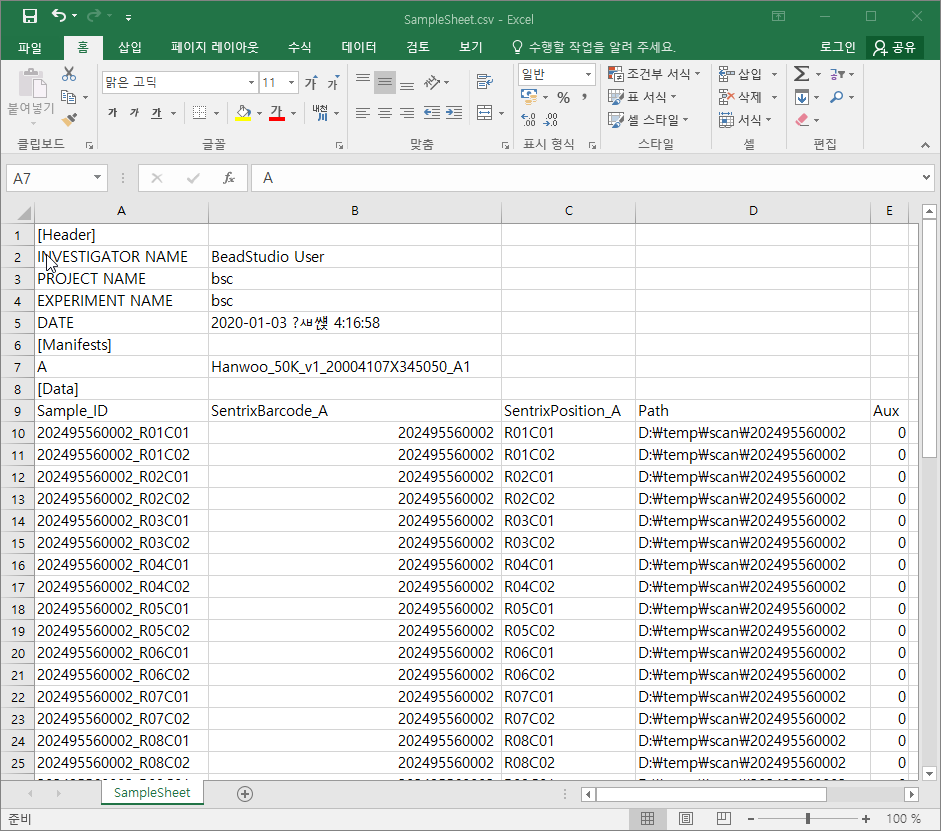
SentrixBarcode_A에 비드칩의 고유번호가 있고, SentrixPosition_A에 비드칩에서의 위치가 나와 있다. Sample_ID에 해당 칩, 해당 위치에서 genotyping한 개체 번호를 입력하고, Path, Aux열을 삭제해도 된다.

csv 파일로 저장한다.
저장한 SampleSheet.csv를 지놈스튜디오에 적용해야 하는데 잘 모르겠다. 이제 완전한 지놈스튜디오 파일을 만들기 위한 모든 게 갖추었다. 비드칩 스캔 파일, egt 파일, bpm 파일, SampleSheet 파일 등 4 종류의 파일을 모두 갖추었다. 그래서 genotyping을 하고 나면 이렇게 4종류의 파일을 모두 보관하기 바란다. 그래야 지놈스튜디오 파일을 만들 수 있으니까.
나는 다음과 같이 보관하고 있다.
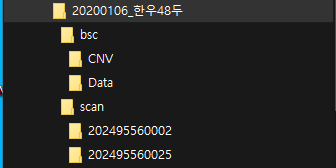
scan 폴더에는 다음과 같이 스캔한 비드칩 폴더와 SampleSheet 파일, bpm 파일, egt 파일을 저장해 나중에라도 지놈스튜디오 파일을 다시 만들 수 있게 했다.
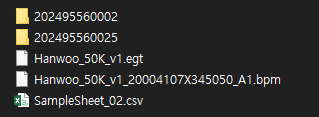
이렇게 한 이유는 지놈스튜디오란 프로그램이 버전업을 하는데 호환성이 없이 버전업을 한 경우가 있었다. 또는 32bit 윈도우즈에서 64bit 윈도우즈로 업그레이드를 하니까 지놈스튜디오 파일이 호환되지 않는 경우가 발생했다. 그래서 나중에라도 지놈스튜디오 파일을 만들 수 있는 재료를 모두 보관하고 있는 것이 맞을 것 같다.
다시 지놈스튜디오 파일을 만들어 보자. 그리고 보통은 Genotyping을 한 실험실에 요구를 하면 스캔한 파일, SampleSheet, egt, bpm 파일을 모두 제공해 준다. GenomeStudio를 실행한다. File – New Project - Genotyping을 클릭한다.

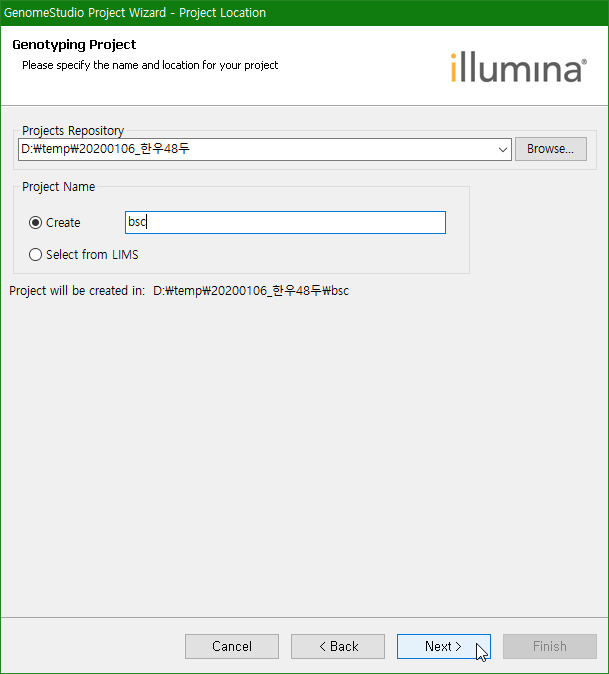
프로젝트를 저장할 폴더를 지정하고 프로젝트 이름을 입력한다. Next를 클릭한다.
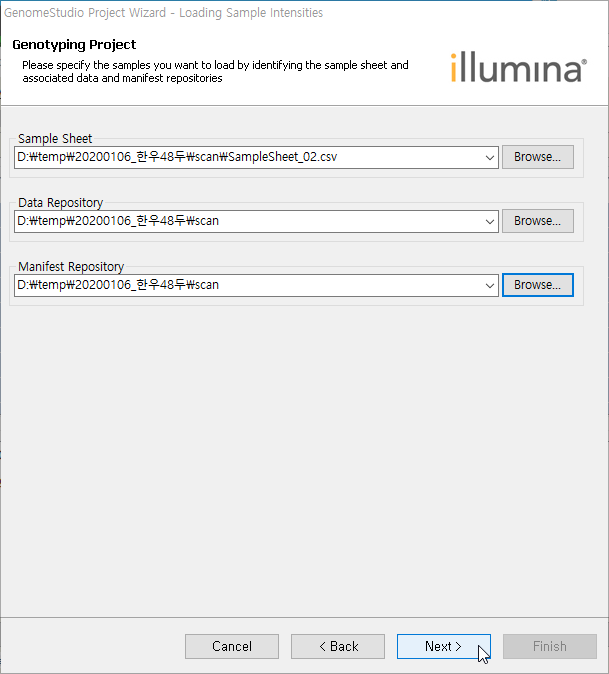
SampleSheet 파일을 지정하고, 비드칩 스캔 폴더가 있는 폴더와 bmp 파일이 있는 폴더를 지정하고 Next를 클릭한다.

Import cluster positions from a cluster file에 체크를 하고 egt 파일을 지정한다. Pre-Calculate와 Calculate Sample and SNP Statistic에 체크한다. Finish를 클릭한다.

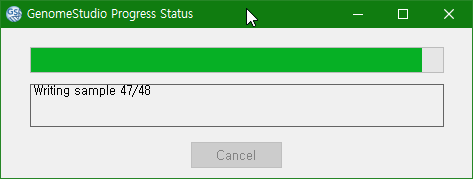

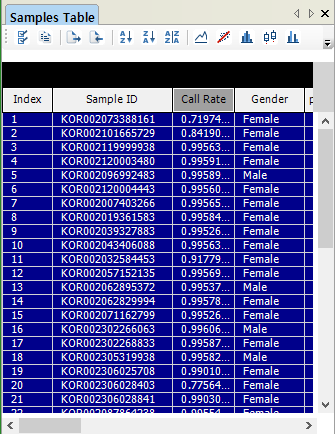
클러스터링이 적용되어 genotype이 결정된 것을 알 수 있고, 왼쪽 아래 창에서 개체의 Call Rate도 확인할 수 있다. 화면에서 Call Rate가 0.71, 0.84를 볼 수 있는데 90% 이하인 개체는 빼는 게 정신 건강에 좋을 것 같다. imputation하면 될 줄 알았는데 잘 안 된다. imputation을 해도 엉뚱하게 된다. 결국 전체 시스템을 망가뜨린다. 10만 원 아까워서 수 천만 원 날리는 일이 없기를 바란다. Call Rate가 낮은 개체들을 SampleSheet에서 지우고 다시 지놈스튜디오 파일을 만들면 읽지 않는다. 실험실에서는 이런 식으로 여러 의뢰자의 샘플을 한 비드칩에 넣어 실험하고, SampleSheet를 조절하여 각 의뢰자의 것만 읽어 지놈스튜디오 파일을 만든다.
[2020.8.15. 추가 시작]
Call Rate가 낮은 개체를 SampleSheet에서 지우고 다시 지놈스튜디오 파일을 만들었는데 다음과 같은 에러가 발생했다.
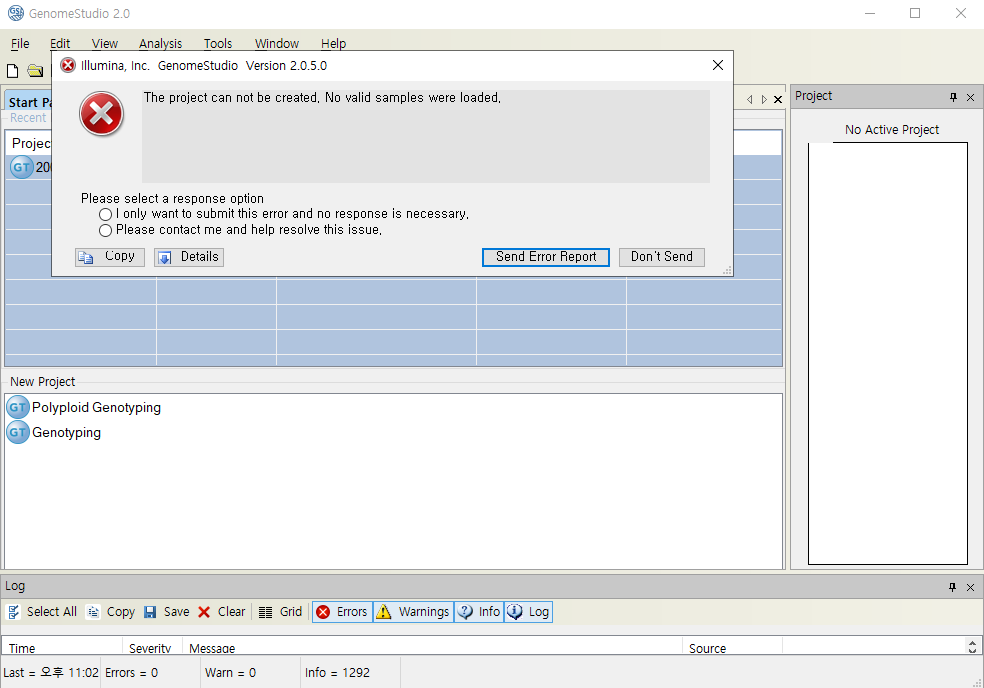
그건 SampleSheet를 다룰 때 문제가 발생했기 때문이다. SampleSheet는 csv 파일인데 이건 사실 엑셀과 상관 없는 파일 형식인데 윈도우즈에서는 이걸 더블 클릭하면 엑셀이 실행되고 다음과 같이 나온다.
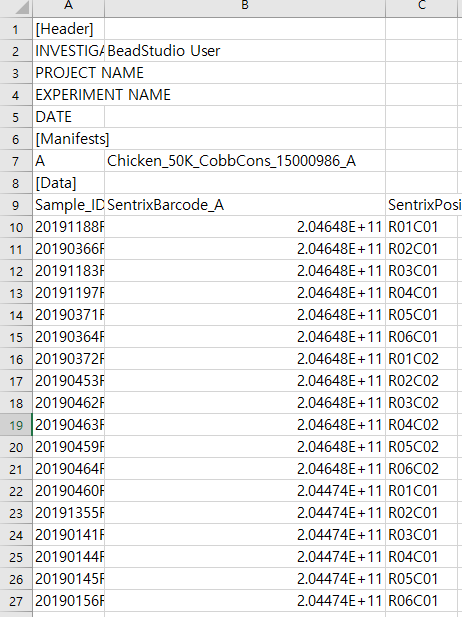
이상한 점을 발견했는가. 칩 번호가 지수로 표시되고 있다. 칩 번호는 사실 숫자가 아니라 숫자로 된 문자열인데 엑셀은 그걸 모른다. 셀 서식을 지정해서 다음과 같이 나오게 한다.
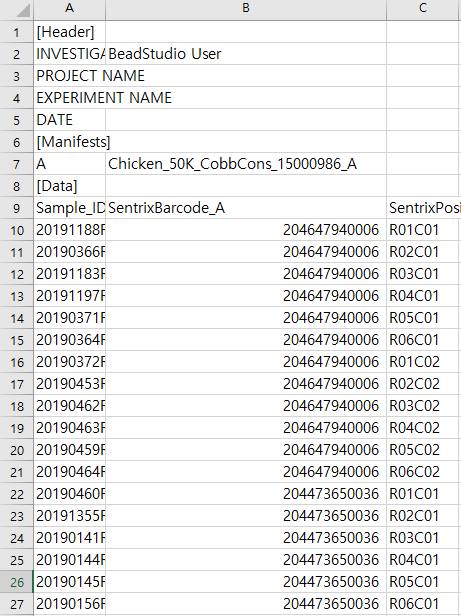
위와 같이 해 놓고 csv 파일로 저장을 해야 한다. csv 파일을 만들었으면 반드시 텍스트 에디터로 확인을 해봐야 한다. SampleSheet를 조작할 때 엑셀이 아니라 텍스트 에디터로 조작하는 것도 방법이다. 최근 기사를 보니 유전자 이름 'MARCH1' 있는데 유전학자들이 이걸 엑셀에 입력을 하면 자꾸 3월 1일로 인식을 해서 골탕을 먹는다는 기사가 있었다. 2004년부터 제기된 문제인데 결국 유전학자들이 'MARCH1' 유전자의 이름을 'MARCHF1'으로 바꾸기로 했다고 한다. 이 정도의 문제는 아닌데 비슷한 류의 문제가 아닌가 싶다. 항상 중간 중간에 체크를 해야 한다. csv 파일을 만들었으면 그걸 엑셀이 아니라 텍스트 에디터로 열어서 잘 만들어 졌는지 확인해야 한다. 모든 자료 분석 시 단계 단계 마다 이런식으로 확인을 해야 한다.
[2020.8.15. 추가 끝]
GenomeStudio에서는 genotype을 보고 gender를 결정할 수 있다. X, Y 염색체의 SNP genotype의 homo와 hetero 비율을 보고 결정한다. 전체 개체를 선택한다.

마우스 오른쪽 버튼 클릭 - Estimate Gender for Selected Samples... 클릭한다.

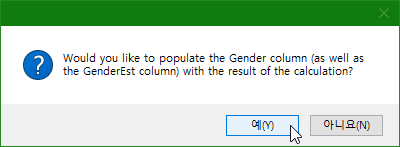
이렇게 gender를 결정하고 이것을 실험과정 중에 샘플이 바뀌었는지 즉 실험이 정확히 되었는지 체크하는 체크 포인트로 사용할 수 있다.
이렇게 bsc 파일을 만들어 보는 것을 해 봤는데, 이걸 꼭 알아야 하나라는 생각이 든다. 왜 필요한지 생각해 보자. 예를 들어 120두씩 매월 분석을 한다고 가정해 보자. 10개월 해서 1200두를 분석했다. 그런데 다음에 알아 볼 finalreport가 필요하다고 했을 때 10번 finalreport 출력하는 작업을 해야 하나? 힘들다. 차라리 한 번 노력해서 1200두 한 번에 뭉쳐서 관리하면 한 방에 1200두 finalreport를 출력하는 것이 편리하지 않은가? 또 소의 경우 BovineSNP50v2가 종료되고 v3가 나오는데 v2가 종료되었으니 여기 저기 찢어져 있는 거 관리하기도 힘드니 하나로 뭉쳐 놓고 싶을 때가 있다. 이럴 때 지놈스튜디오 파일 만드는 방법을 알아야 한다. 위에서도 설명을 했듯이 지놈스튜디오가 버전업 되었을 때 혹시 호환성이 사라지면 새로 지놈스튜디오 파일을 만들어야 할 때도 있다. 암튼 알면 좋을 것 같다. 모르면 위에서 얘기해 놨듯이 분석했을 때 genotype 자료만 받지 말고 스캔한 이미지 파일, SampleSheet 파일, egt 파일, bpm 파일을 다 받아서 저장해 두자. 그래야 누구한테 부탁을 해도 할 것이 아닌가. 실제 몇 년 전에 국내에서 SNP genotyping 회사가 문을 닫으면서 그 회사가 분석했던 자료가 다 사라졌다. 그런데 finalreport를 다른 형태로 다운로드 받아 보고 싶었지만, 지놈스튜디오 파일도 없고, 스캔한 이미지 파일도 없어서 수 천 두의 자료를 이용하지 못한 일도 있었다(그때는 1두 분석비가 20 ~ 30만 원이었다).
'Animal Breeding > Genomic Selection' 카테고리의 다른 글
| QCF90이용한 quality control (0) | 2020.02.05 |
|---|---|
| PREGSF90을 이용한 Quality Control (0) | 2020.02.04 |
| FImpute를 이용한 SNP Imputation (1) | 2020.02.03 |
| illumina2pregs로 SNP genotype을 0, 1, 2로 변환하기 (0) | 2020.01.09 |
| GenomeStudio FinalReport 만들기 (1) | 2020.01.08 |