육종을 하면서 집단에 있는 개체들의 근교계수를 계속 계산하는 것은 매우 중요한 일이다. 근친교배(inbreeding)가 일어나면 근교퇴화(inbreeding depression)가 발생하기 때문이다. 근교퇴화란 치사 유전자가 발현하여 죽어 버리거나 죽지 않더라도 경제 능력이 감소하는 현상을 말한다. 근교계수를 계산해 주는 수많은 프로그램들이 있지만 오늘은 inbupgf90을 이용하여 근교계수를 계산해 보자.
inbupgf90을 다운로드 하자.
http://nce.ads.uga.edu/html/projects/programs/Windows/64bit/
Index of /html/projects/programs/Windows/64bit
nce.ads.uga.edu
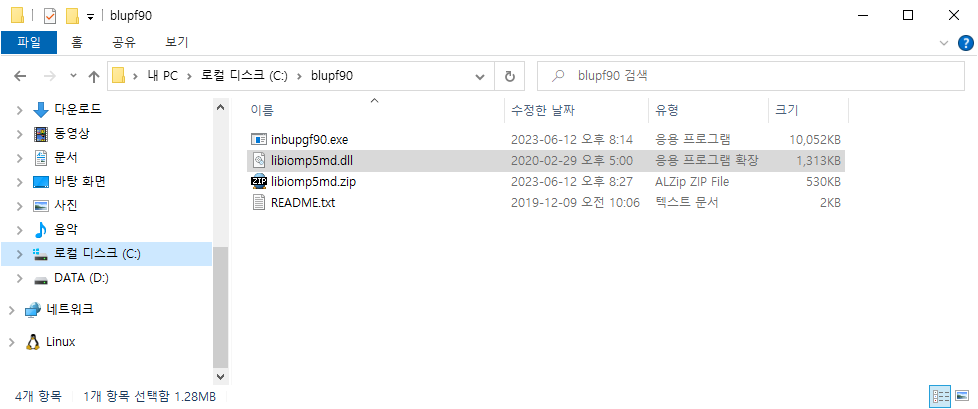
c:\ 밑에 blupf90이라는 폴더를 만들고 다운로드 받은 inbupgf90을 복사한다.

그러나 이 파일만으로는 실행이 안된다. 라이브러리 파일이 필요하다. libiomp5md.dll이라는 파일이 필요하다.
검색을 해서 다운로드 받거나 다음 링크를 연결하여 다운로드 한다.
https://ko.dll-files.com/libiomp5md.dll.html
libiomp5md.dll 무료 다운로드 | DLL‑files.com
libiomp5md.dll, 파일 설명: Intel(R) OMP Runtime Library libiomp5md.dll와(과) 관련된 오류는 몇 가지 다른 이유로 발생할 수 있습니다. 예를 들어, 응용 프로그램에 결함이 있거나, libiomp5md.dll이(가) PC에 존재하
ko.dll-files.com
다운로드하고 압축을 푼 결과
명령 프롬프트 실행 : 윈도우 시작 -> 찾기 창에서 cmd 입력

cd .. 명령어로 상위 폴더로 이동
cd .. 명령어로 한번 더 상위 폴더로 이동
cd blupf90으로 blupf90 폴더로 이동
dir 명령어로 폴더 내의 파일 확인

inbupgf90 명령어로 프로그램 실행

설치가 잘 된 것을 확인할 수 있다.마지막으로 모든 폴더에서 실행이 가능하도록 path를 설정하자.윈도우 시작 -> 설정 -> 정보 -> 고급 시스템 설정


환경 변수 클릭
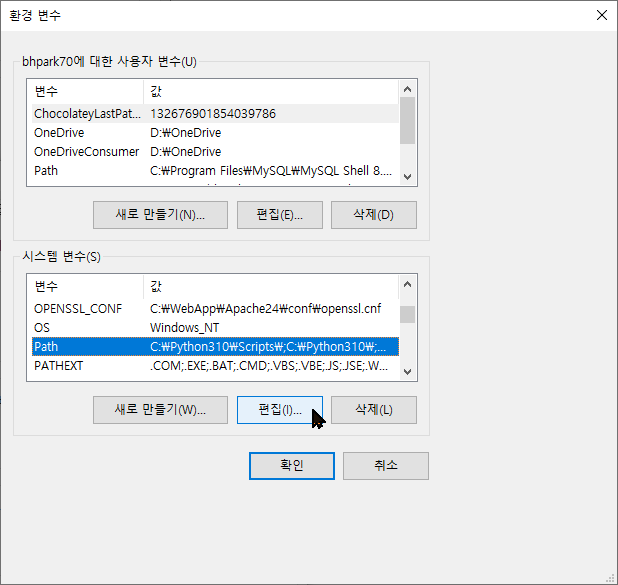
시스템 변수 -> Path -> 편집 클릭

새로 만들기 클릭

C:\blupf90 입력하고 확인명령 프롬프트를 다시 실행하면 어떤 폴더에서든지 inbupgf90을 실행할 수 있다.

지금까지 프로그램을 실행하기 위한 환경 설정이었다. 한가지 더. D: 드라이브로 이동하기 위해서는 D: 명령어를 입력한다.

다시 기억해 보자. 위로 올라가는 명령어는 "cd .." 그리고 폴더로 이동하는 명령어는 "cd 폴더이름"이다.
이제 자료를 준비하고 프로그램을 실행하여 근교계수를 계산해 보자. 그리고 미래에 태어날 개체의 근교계수를 계산해 보자.
근교 계수를 구할 개체들의 혈통 자료 준비

여기서는 12422두를 준비하였다. 부모를 모를 경우 0으로 기입한다.
여기서 개체, 아비 및 어미 사이를 탭을 사용하여 구분하지 말고, 스페이스를 사용하여 구분한다. 엑셀에서 copy & paste를 할 경우 탭으로 구분된 경우가 있다. 이럴 경우 탭을 복사해서 찾아서 바꾸기를 할 수도 있고, 에디터에서 탭을 스페이스로 변환할 수 있다. 각자 편한 방법으로.
다음과 같은 간단한 명령어로 근교계수 계산이 끝난다.
inbupgf90 --pedfile kbh_pedi_ori_202305.txt --method 3 | tee inbupgf90_01.log
--pedfile 뒤에 혈통 파일의 이름을 적어 준다.
근교계수 계산 방법을 지정할 수 있다. 1번과 2번은 Aguilar & Misztal, 2008의 방법을 이용하고 3번은 Meuwissen & Luo 1992의 방법을 이용한다. --method 옵션을 생략하면 2번 방법을 이용하는데 메모리에서 계산하여 빠르지만 많은 메모리를 요구한다. 그러나 그렇게 많은 혈통이 있나 모르겠다. 그냥 생략해도 된다.
| tee inbupgf90_01.log 는 inbupgf90_01.log 파일에 화면에 나오는 정보를 저장하고 나중에 천천히 보기 위함이다.
그런데 이 tee 명령이 실행될려면 윈도우에 프로그램을 설치해야 하는데 그건 다음 포스팅을 참조한다. 복잡하다고 느낀다면 " | tee inbupgf90_01.log" 없이 실행한다.
2009.03.17 - [ETC] - GnuWin32의 CoreUtils 패키지 설치하기
GnuWin32의 CoreUtils 패키지 설치하기
윈도우즈에서 유닉스 명령어 사용하기GnuWin32가 있음http://gnuwin32.sourceforge.net/ GnuWin32에는 여러 패키지가 있는데 CoreUtils에 내가 필요한 sort 프로그램이 있음http://www.gnu.org/software/coreutils/ CoreUtils Dow
bhpark.tistory.com
실행 화면


25% 초과가 4마리 있다.
마지막에 근교계수가 저장된 파일의 이름이 나온다.
계산된 근교계수 확인

근교계수가 0이라고 해서 정말 근친교배가 아닌 것은 아니다. 혈통에 기반한 것이기 때문에 혈통이 부족하면 0이 나온다. 그래서 혈통 완전도 지수(pedigree complete index)를 보고 근교계수를 판단해야 한다.
2015.11.24 - [Animal Breeding/Animal Breeding Etc] - Pedigree Completeness Index
Pedigree Completeness Index
Pedig의 ngen에서 만들어진 결과 파일을 이용하여 혈통완성도지수(Pedigree Completeness Index)를 계산한다. Pedigree completeness index란 ○ 지정한 세대까지 혈통이 얼마나 완전한지를 나타내는 지수 ○ 혈통
bhpark.tistory.com
근교계수를 계산했는데 태어난 개체의 근교계수를 계산하면 뭐하나. 태어나기 전에 계산을 해서 근교계수가 높을 것 같으면 그런 교배를 회피해야 한다. 그래서 inbupgf90에서는 미래에 태어날 개체의 근교계수를 계산해 준다. 교배에 사용할 아비와 어미 목록을 준비하면 각각의 아비와 각각의 어미가 교배하여 태어날 개체의 근교계수를 알려준다.
먼저 교배에 사용할 아비 목록을 준비한다.

교배에 사용할 어미 목록을 준비한다.
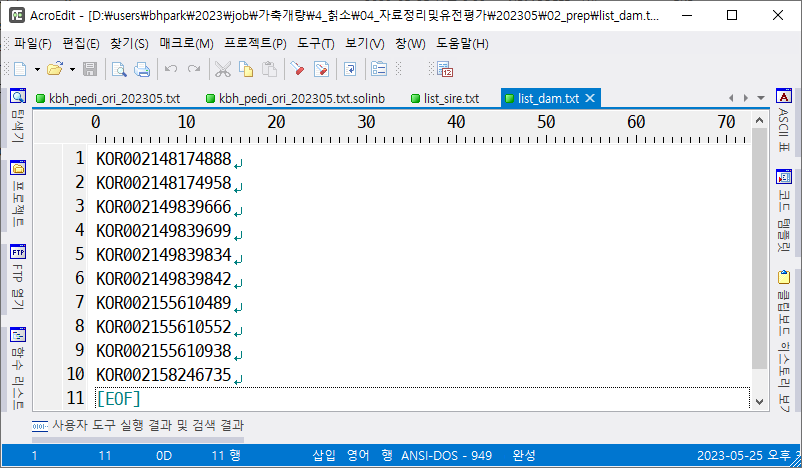
다음과 같이 명령어를 실행한다.
inbupgf90 --pedfile kbh_pedi_ori_202305.txt --sire_file list_sire.txt --dam_file list_dam.txt --matings | tee inbupgf90_01.log
--sire_file 준비한 아비 목록이 담긴 파일 이름
--dam_file 준비한 어미 목록이 담긴 파일 이름
--matings 태어날 개체의 근교계수 계산
실행 화면

여러 파일이 생기는데 .matings 파일을 보자.
DamID SireId_MinInb KOR002154583225 KOR002154584201 KOR002154584156 KOR002154584130 KOR002154583321 KOR002166240033 KOR002160500515 KOR002166240308 KOR002154584148 KOR002166240381
KOR002149839834 KOR002154583225 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000
KOR002148174888 KOR002154584201 3.906 0.781 4.688 4.688 1.562 3.906 8.594 5.078 5.469 16.797
KOR002155610552 KOR002154583225 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000 0.000
KOR002155610489 KOR002154584201 1.172 0.391 2.344 1.562 0.781 1.172 1.172 0.977 1.172 1.367
KOR002148174958 KOR002154583225 0.000 0.391 0.781 0.195 0.000 0.391 0.391 0.195 0.000 0.195
KOR002155610938 KOR002154584201 1.855 0.879 7.617 2.539 1.758 1.465 1.660 1.416 1.660 1.709
KOR002149839699 KOR002154584201 1.172 0.391 2.344 1.562 0.781 1.172 1.172 0.977 1.172 1.367
KOR002149839842 KOR002154584201 3.320 1.172 2.734 2.930 2.344 1.953 1.758 2.148 2.539 2.148
KOR002158246735 KOR002154584201 1.562 0.781 7.031 1.953 1.562 1.172 1.367 1.172 1.172 1.367
KOR002149839666 KOR002154584201 1.172 0.391 2.344 1.562 0.781 1.172 1.172 0.977 1.172 1.367어미별로 각 아비와 교배했을 때 태어날 개체의 근교계수를 계산해 주고, 그 중 가장 근교계수를 보인 아비를 표시해 준다. 그러나 그 아비의 근교계수만 최소인 것은 아니다. 다른 아비와 교배해도 0인 경우가 많다. 보고 고르면 된다.
_relationships 파일은 아비 사이의 혈연 계수를 보여준다.
KOR002154583225 KOR002154583225 1.047
KOR002154583225 KOR002154584201 0.105
KOR002154583225 KOR002154584156 0.086
KOR002154583225 KOR002154584130 0.133
KOR002154583225 KOR002154583321 0.180
KOR002154583225 KOR002166240033 0.285
KOR002154583225 KOR002160500515 0.090
KOR002154583225 KOR002166240308 0.111
KOR002154583225 KOR002154584148 0.129
KOR002154583225 KOR002166240381 0.078
KOR002154584201 KOR002154584201 1.000
KOR002154584201 KOR002154584156 0.047
KOR002154584201 KOR002154584130 0.080
KOR002154584201 KOR002154583321 0.195
KOR002154584201 KOR002166240033 0.125
KOR002154584201 KOR002160500515 0.172
KOR002154584201 KOR002166240308 0.273
KOR002154584201 KOR002154584148 0.152
KOR002154584201 KOR002166240381 0.086
KOR002154584156 KOR002154584156 1.062
KOR002154584156 KOR002154584130 0.125
KOR002154584156 KOR002154583321 0.078
KOR002154584156 KOR002166240033 0.078
KOR002154584156 KOR002160500515 0.195
KOR002154584156 KOR002166240308 0.070
KOR002154584156 KOR002154584148 0.070
KOR002154584156 KOR002166240381 0.141
KOR002154584130 KOR002154584130 1.031
KOR002154584130 KOR002154583321 0.156
KOR002154584130 KOR002166240033 0.072
KOR002154584130 KOR002160500515 0.090
KOR002154584130 KOR002166240308 0.087
KOR002154584130 KOR002154584148 0.305
KOR002154584130 KOR002166240381 0.074
KOR002154583321 KOR002154583321 1.000
KOR002154583321 KOR002166240033 0.086
KOR002154583321 KOR002160500515 0.117
KOR002154583321 KOR002166240308 0.113
KOR002154583321 KOR002154584148 0.086
KOR002154583321 KOR002166240381 0.043
KOR002166240033 KOR002166240033 1.016
KOR002166240033 KOR002160500515 0.070
KOR002166240033 KOR002166240308 0.117
KOR002166240033 KOR002154584148 0.121
KOR002166240033 KOR002166240381 0.082
KOR002160500515 KOR002160500515 1.016
KOR002160500515 KOR002166240308 0.133
KOR002160500515 KOR002154584148 0.117
KOR002160500515 KOR002166240381 0.293
KOR002166240308 KOR002166240308 1.023
KOR002166240308 KOR002154584148 0.154
KOR002166240308 KOR002166240381 0.223
KOR002154584148 KOR002154584148 1.016
KOR002154584148 KOR002166240381 0.123
KOR002166240381 KOR002166240381 1.055
_statistics 파일은 아비를 기준으로 평균, 최소, 최대 근교계수를 보여 준다.
1 KOR002154583225 1.416 1.172 3.906 3
2 KOR002154584201 0.518 0.391 1.172 7
3 KOR002154584156 2.988 0.781 7.617 0
4 KOR002154584130 1.699 0.195 4.688 0
5 KOR002154583321 0.957 0.781 2.344 0
6 KOR002166240033 1.240 0.391 3.906 0
7 KOR002160500515 1.729 0.391 8.594 0
8 KOR002166240308 1.294 0.195 5.078 0
9 KOR002154584148 1.436 1.172 5.469 0
10 KOR002166240381 2.632 0.195 16.797 0마지막 열의 수는 _matings 파일에 추천 아비로 몇 번 되었는지를 나타내는 수이다.
교배계획을 어떻게 할 것인가? 근교계수가 최소로 상승하면서 최대의 능력을 발휘하는 교배가 좋은 교배이긴 하겠지만 그게 쉽지 않다. 어떤 아비가 집단의 어미와 근교도 안 되고 능력도 최고치로 올린다고 해서 모든 어미에 교배하면 그 다음 세대에 곤란한 일이 발생할 것이다. 이와 관련된 문제를 멋지게 풀어 보려고 Optimal Contribution Selection이란 개념도 생긴 거 같다.
사용 가능한 아비를 골라 보고, 어미를 기준으로 어느 정도 이하의 근교계수를 보이는 아비들을 골라보고 그 아비 중 최대의 능력을 보이는 아비를 골라보자. 그리고 전체적으로 골고루 골라졌나 보자. 한 두 개의 아비로 몰렸다면 적당히 조정하자.
'Animal Breeding > BLUPF90' 카테고리의 다른 글
| blupf90으로 social interaction model 풀기 (1) | 2021.04.21 |
|---|---|
| blupf90으로 single-step GBLUP Model 풀기 (0) | 2021.01.05 |
| blupf90으로 GBLUP Model with residual polygenic effects 풀기 (0) | 2020.12.29 |
| blupf90으로 gblup model 풀기 (0) | 2020.12.29 |
| blupf90으로 SNP-BLUP Model(with polygenic effects unweighted analysis) 풀기 (0) | 2020.12.24 |